코딩하는 해맑은 거북이
[데이터분석] 건강검진 데이터로 가설 검정하기 본문
본 게시물의 내용은 '[코칭스터디 13기] Data Science 2023 → 파이썬으로 시작하는 데이터 사이언스(박조은)' 강의를 듣고 작성하였다.
해당 글은 아래의 내용을 다룬다.
📢 사용 데이터셋 정보
💡 데이터 로드하기
💡 데이터 미리보기
💡 데이터 요약정보 살펴보기
💡 결측치 확인하기
💡 일부 데이터 기초통계값 확인하기
💡 일부 데이터 값 집계하기
✔️ value_counts
✔️ groupby
✔️ pivot_table
💡 전체 데이터 시각화 하기
✔️ hist - 히스토그램
💡 샘플 데이터 시각화 하기
🪄 샘플 데이터 추출하기
🪄 범주형 데이터 시각화
✔️ countplot
✔️ barplot
✔️ lineplot
✔️ pointplot
✔️ boxplot
✔️ violinplot
✔️ swarmplot
🪄 수치형 데이터 시각화
✔️ scatterplot
✔️ lmplot
✔️ distplot
✔️ kdeplot
💡 상관 분석
✔️ corr - 상관계수 구하기
✔️ heatmap
공공데이터 포털의 ' 국민건강보험공단_건강검진정보'의 '건강검진정보(2017).csv' 사용
https://www.data.go.kr/data/15007122/fileData.do
건강검진정보란 국민건강보험의 직장가입자와 40세 이상의 피부양자, 세대주인 지역가입자와 40세 이상의 지역가입자의 일반건강검진 결과와 이들 일반건강검진 대상자 중에 만40세와 만66세에 도달한 이들이 받게 되는 생애전환기건강진단 수검이력이 있는 각 연도별 수진자 100만 명에 대한 기본정보(성, 연령대, 시도코드 등)와 검진내역(신장, 체중, 총콜레스테롤, 혈색소 등)으로 구성된 개방데이터이다.
여기서 가설은 '음주여부 혹은 흡연상태에 따라 건강검진 수치 차이가 있을까?'로 세우고 분석을 진행해본다!
shape를 통해 1000000개의 행과 34개의 열로 이루어진 것을 확인할 수 있다.
df = pd.read_csv("data/NHIS_OPEN_GJ_2017.CSV", encoding="cp949")
df.shape
각 컬럼에 대한 정보는 공공데이터 포털에 자세하게 나와있다.
몇 개의 컬럼을 살펴보면, 아래와 같다.


임의로 1개의 데이터를 미리 살펴보자!
* df.head()와 df.tail()을 통해서도 첫 부분과 마지막 부분의 5개의 행씩 살펴볼 수 있다.
df.sample()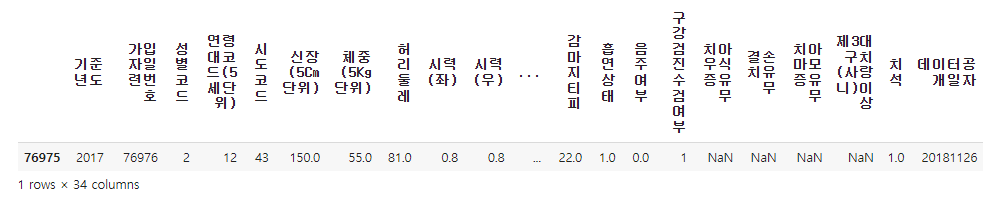
df.info()를 통해 컬럼들(Column)과 각 컬럼들의 데이터 타입(Dtype), 메모리 사용량 등을 확인할 수 있다.
이때, Non-null Count가 총 행의 수와 같다면 결측치가 없는 것이고, 그것보다 적다면 결측치가 있는 것으로 볼 수 있다.
* df.columns : 컬럼명만 출력
* df.dtypes : 데이터 타입만 출력
df.info()
df.isnull()은 데이터프레임을 결측치라면 True, 결측치가 아니라면 False로 출력해주는 함수이다.
이때, sum() 함수를 사용하여 결측치의 개수를 확인할 수 있다.
df.isnull().sum()
# 참고) isna로도 결측치 여부를 확인할 수 있다.
df.isna().sum()
"(혈청지오티)ALT", "(혈청지오티)AST" 컬럼을 가져와서 describe로 요약해보자.
df[["(혈청지오티)ALT", "(혈청지오티)AST"]].head()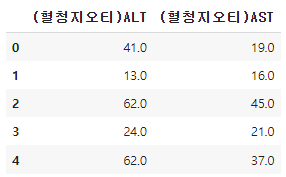
df[["(혈청지오티)ALT", "(혈청지오티)AST"]].describe()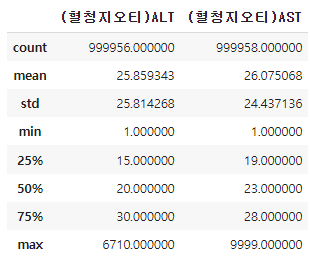
cf) ALT와 AST는 간세포에 들어있는 효소이다. 간이 손상돼 간세포가 파괴되면 그 안에 있던 ALT와 AST가 빠져나와 혈액 속에 섞여 돌아다니게 된다. 따라서 간이 손상되면 ALT와 AST의 수치가 높아진다. 하지만, 간혹 전날 술을 마시거나 몸이 피곤하면 일시적으로 ALT와 AST의 수치가 높아질 수 있어 딱 한 번의 검사만으로 간질환을 판단하는 경우는 드물다고 한다.
"성별코드"와 "흡연상태" 컬럼을 가져와서 그룹화하고 갯수를 집계해보자.
df["성별코드"].value_counts()
df["흡연상태"].value_counts()
groupby를 통해 "성별코드"로 그룹화하여 count 함수로 갯수를 셀 수 있다.
df.groupby(["성별코드"])["가입자일련번호"].count()
여기서 "성별코드"와 "음주여부"로 더 세분화하여 그룹화를 하고 갯수를 세어볼 수 있다.
df.groupby(["성별코드", "음주여부"])["가입자일련번호"].count()
또한, mean 함수로 "감마지피티"의 평균을 구할 수 있다.
df.groupby(["성별코드", "음주여부"])["감마지티피"].mean()
그리고 그룹화한 기초통계값을 describe 함수로 요약정보를 구할 수 있다.
df.groupby(["성별코드", "음주여부"])["감마지티피"].describe()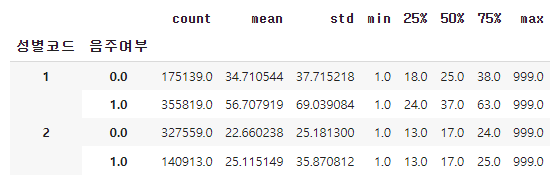
여기서 agg를 사용하면, 구하고자하는 여러 수치를 함께 구할 수 있다.
df.groupby(["성별코드", "음주여부"])["감마지티피"].agg(
["count", "mean", "median"])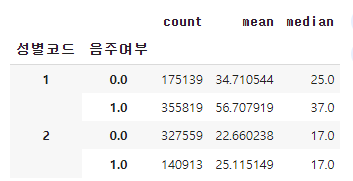
pandas.pivot_table — pandas 2.1.3 documentation
If a list of functions is passed, the resulting pivot table will have hierarchical columns whose top level are the function names (inferred from the function objects themselves). If a dict is passed, the key is column to aggregate and the value is function
pandas.pydata.org
groupby와 비슷하게 pivot_table로도 그룹화하여 구하고자 하는 여러 수치를 구할 수 있다.
pivot_table을 사용하는 방법은 데이터프레임에서 pivot_table을 부르거나, pandas에서 pivot_table을 부를 수 있다.
df.pivot_table(index="음주여부", values="가입자일련번호", aggfunc="count")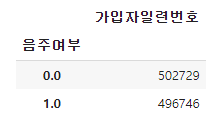
pd.pivot_table(df, index="음주여부", values="감마지티피") # 기본값은 평균(mean)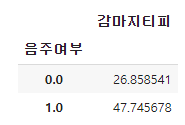
pivot_table의 계산 기본값은 평균이지만, aggfunc를 통해 수치를 지정해줄 수 있다.
pd.pivot_table(df, index="음주여부", values="감마지티피",
aggfunc=["mean", "median"])
aggfunc에 describe를 통해 기초통계값을 확인할 수 있고, index에 여러 컬럼으로 더 세분화도 진행할 수 있다.
pd.pivot_table(df, index="음주여부", values="감마지티피",
aggfunc="describe")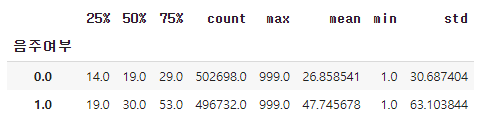
pd.pivot_table(df, index=["성별코드", "음주여부"], values="감마지티피",
aggfunc="describe")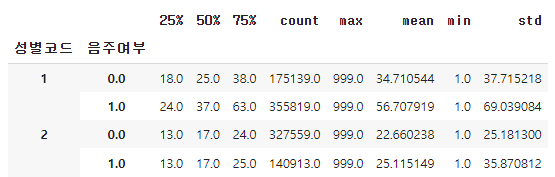
이전에 데이터의 타입을 info로 확인해보았을 때, 대부분 수치 데이터로 이루어진 것을 확인할 수 있었다.
히스토그램을 이용하면 수치데이터를 bin 갯수 만큼 그룹화하여 도수분포표를 만들고 그 결과를 시각화할 수 있다.
h = df.hist(figsize=(16, 16))
여기서 슬라이싱을 이용해서 일부 데이터에 대해서 나누어 그릴 수 있다.
슬라이싱을 사용할 때, iloc[행, 열] 순으로 인덱스를 써주어 데이터를 불러온다.
h = df.iloc[:, :12].hist(figsize=(12, 12))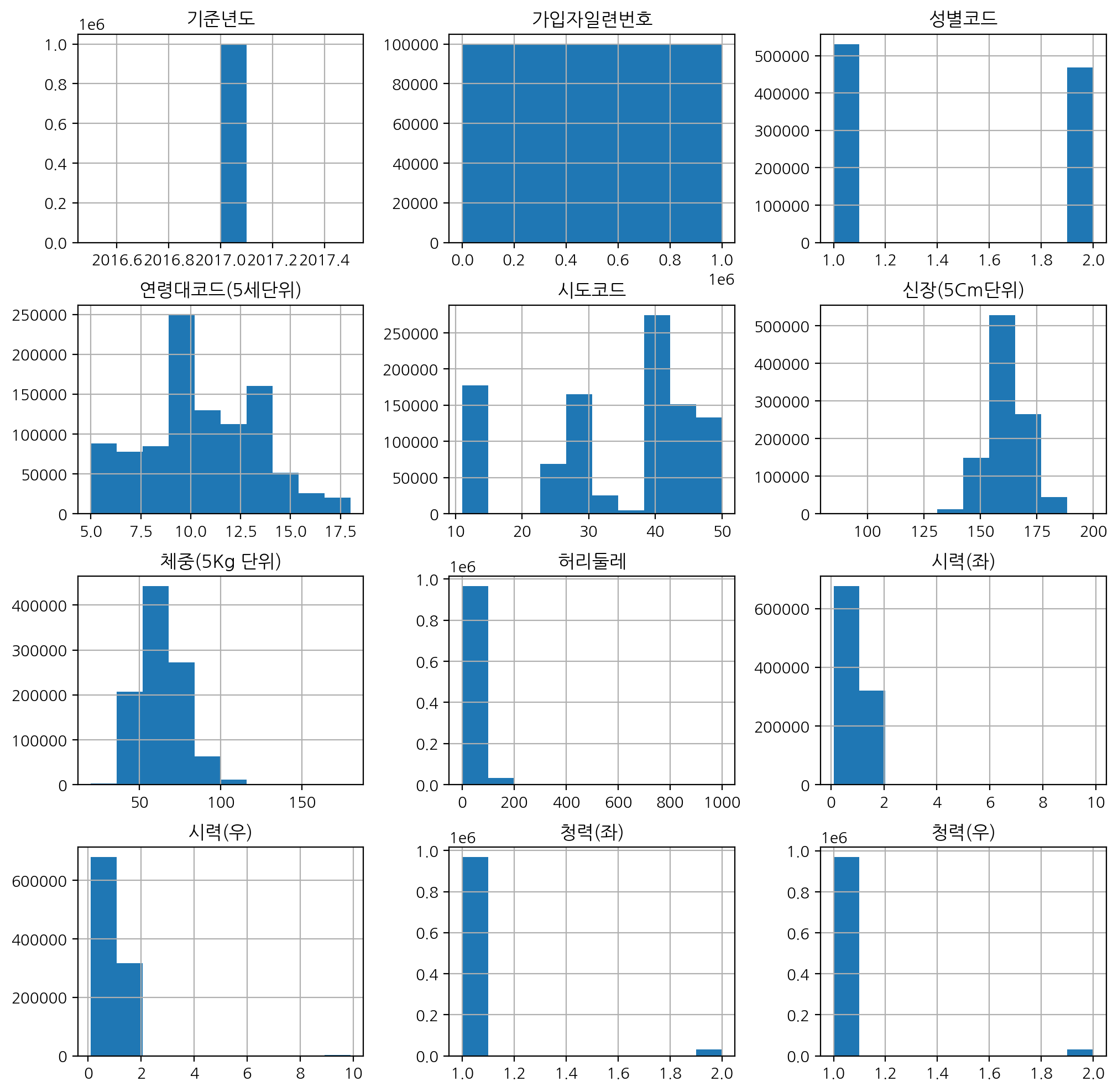
또한, bins로 막대그래프를 몇 구간으로 그릴건지 지정해줄 수 있다.
h = df.iloc[:, 12:24].hist(figsize=(12, 12), bins=100) # bins: 막대그래프를 몇구간으로 그릴건지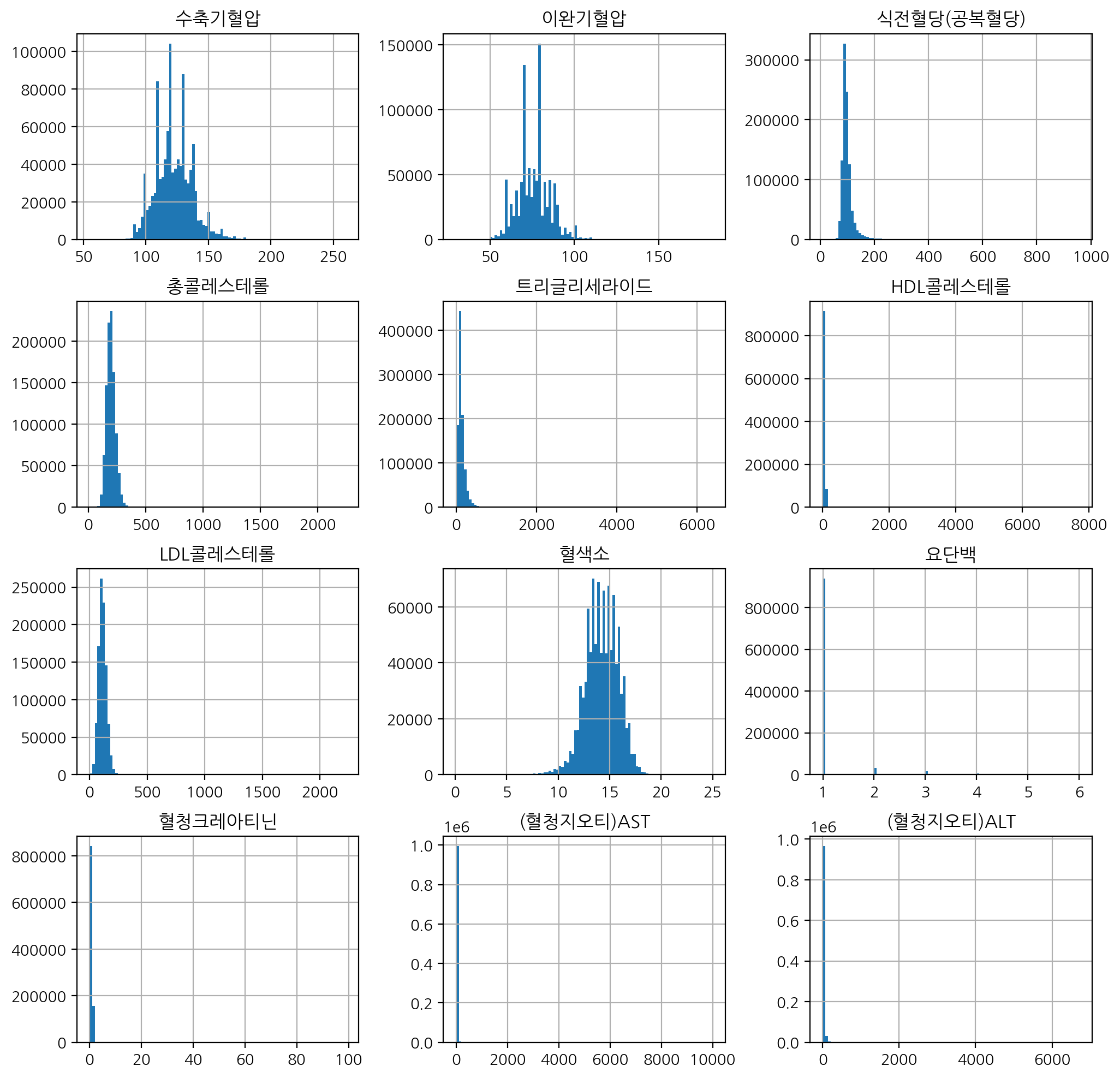
sample 함수를 통해 일부 데이터 몇 개를 지정하여 추출할 수 있다.
여기서 random_state를 고정하여 실험을 통제하기 위해 추출한 데이터를 고정시킬 수 있다.
df_sample = df.sample(1000, random_state=1)
df_sample.shape
아래 시각화는 seaborn을 통해 간단하게 연산해보자! -> import seaborn as sns
seaborn: statistical data visualization — seaborn 0.13.0 documentation
seaborn: statistical data visualization
seaborn.pydata.org
범주형 데이터의 갯수를 그래프로 표현한다.
value_counts()를 구한 값을 시각화한 것과 동일하다.
sns.countplot(x="음주여부", data=df)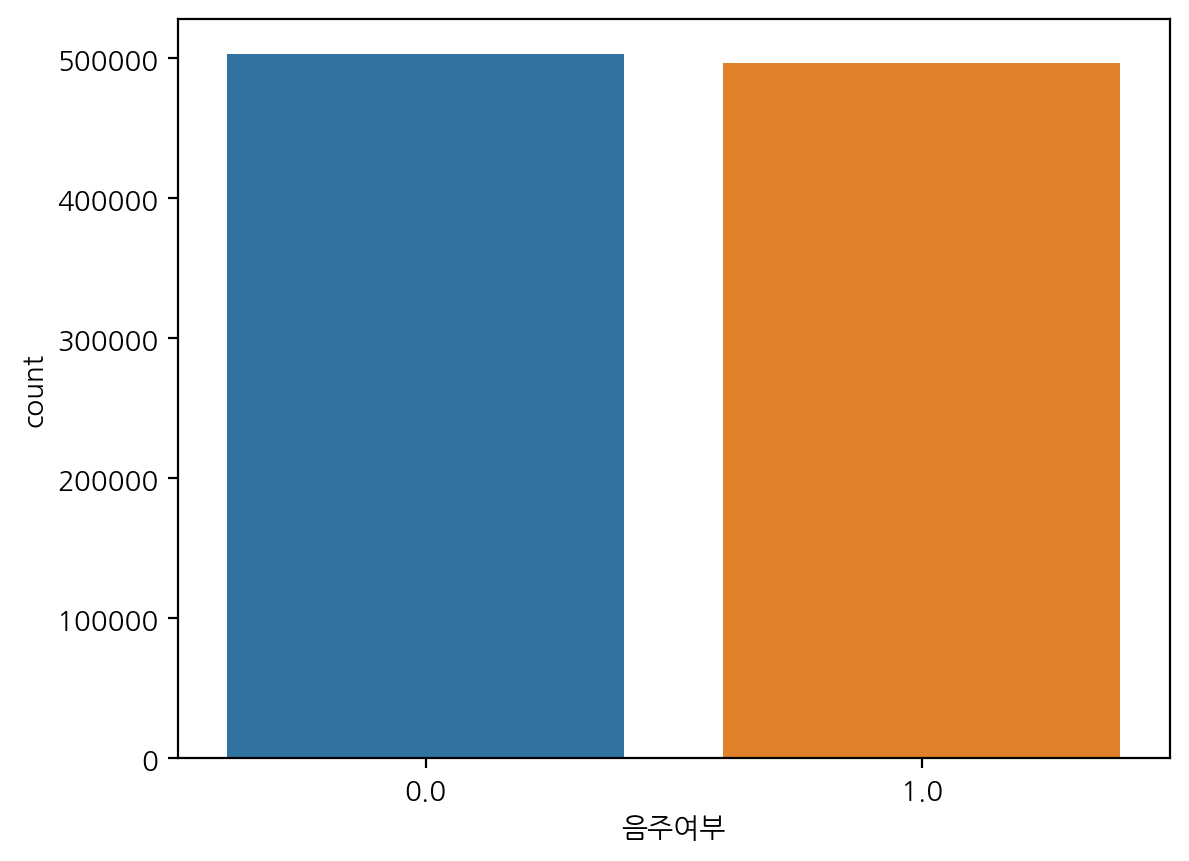
hue 옵션을 사용하여 특정 컬럼에 대해 색상을 구분하여 그릴 수 있다.
sns.countplot(data=df, x="음주여부", hue="성별코드")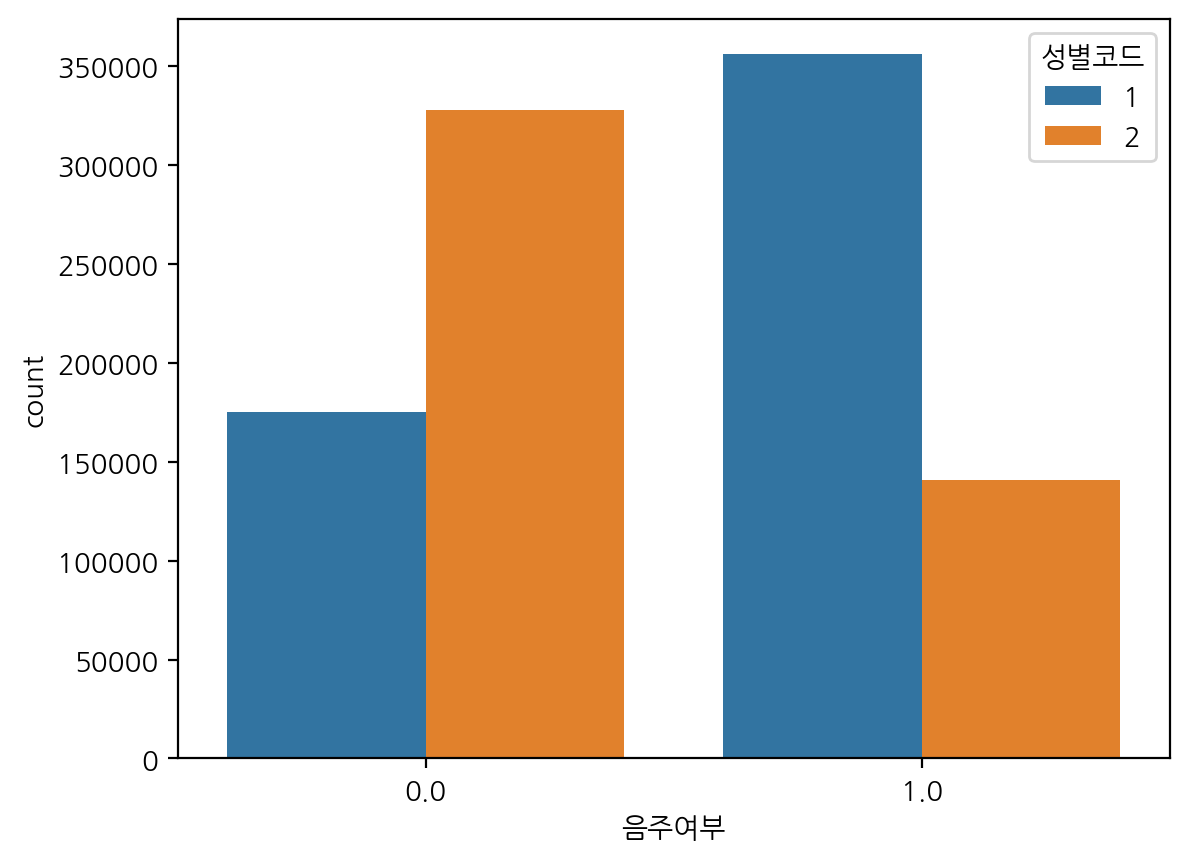
countplot으로 연령대코드별 음주여부를 확인해보자.
sns.countplot(data=df, x="연령대코드(5세단위)", hue="음주여부")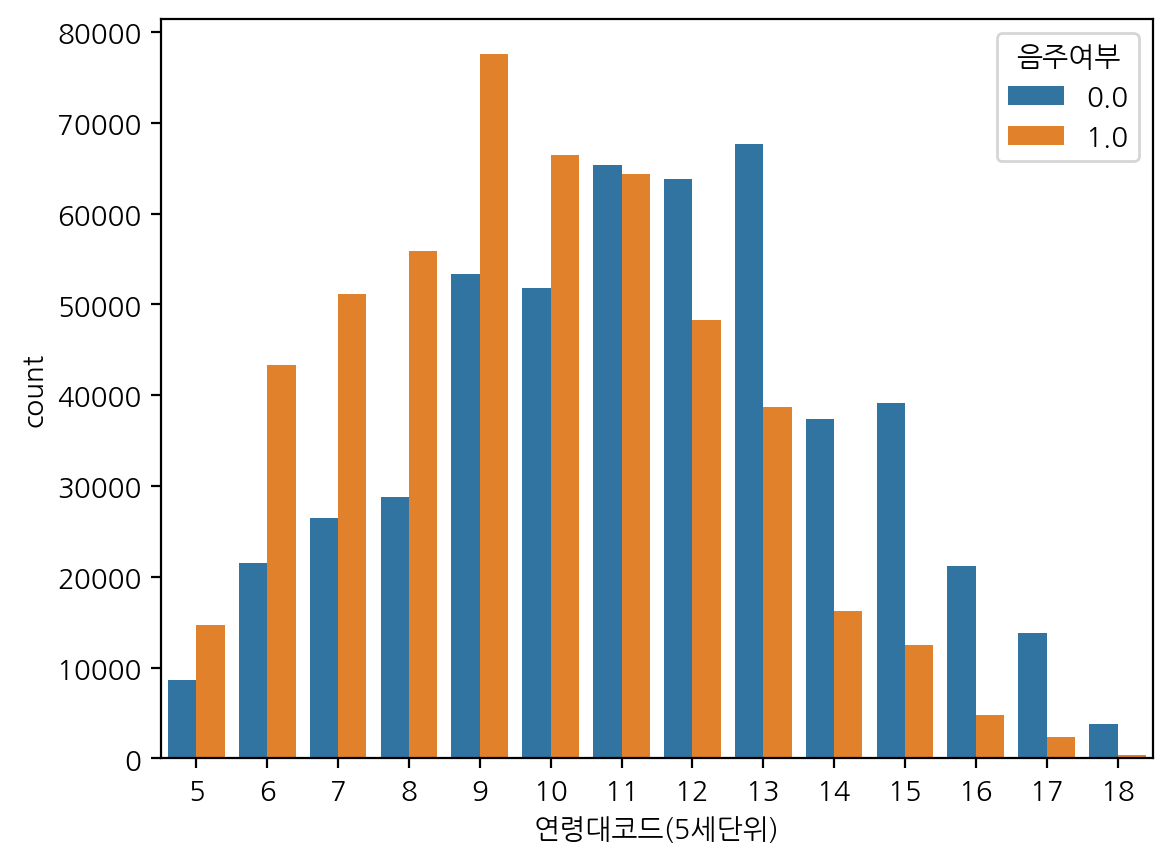
신장과 체중별 갯수도 확인해보자.
plt.figure(figsize=(15, 4))
sns.countplot(data=df, x="신장(5Cm단위)")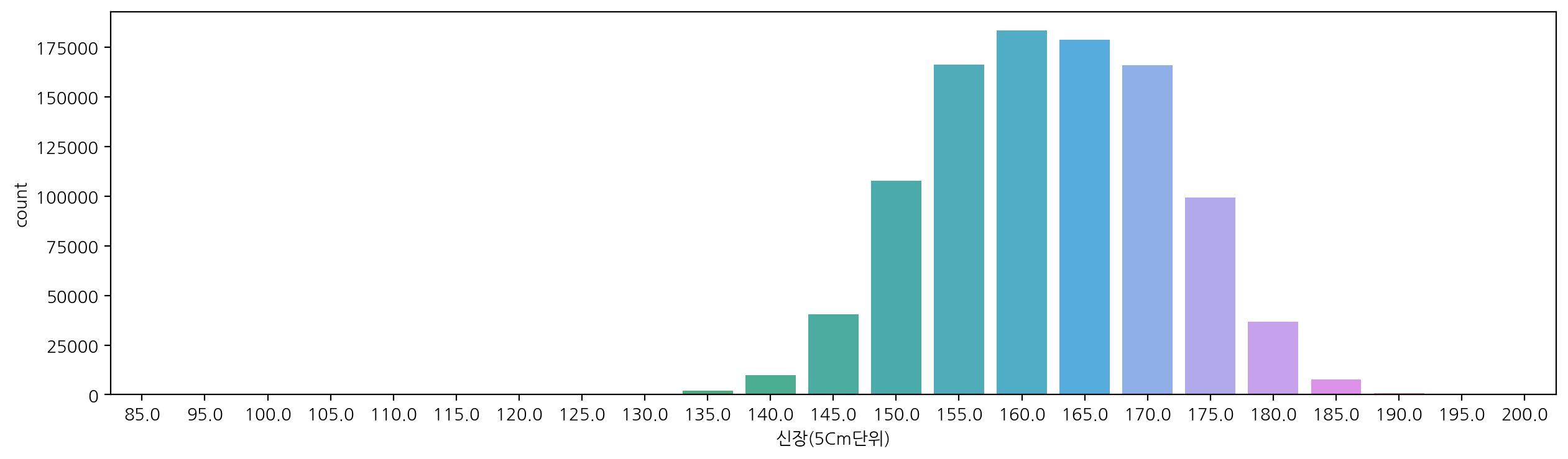
plt.figure(figsize=(15, 4))
sns.countplot(data=df, x="신장(5Cm단위)", hue="성별코드")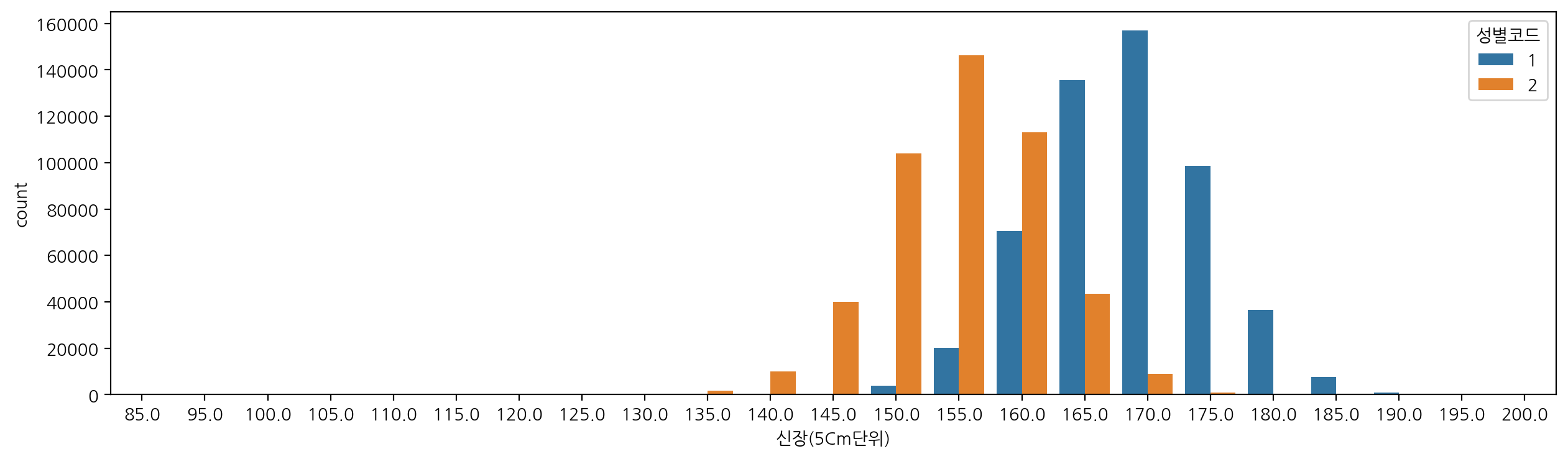
plt.figure(figsize=(15, 4))
sns.countplot(data=df, x="체중(5Kg 단위)")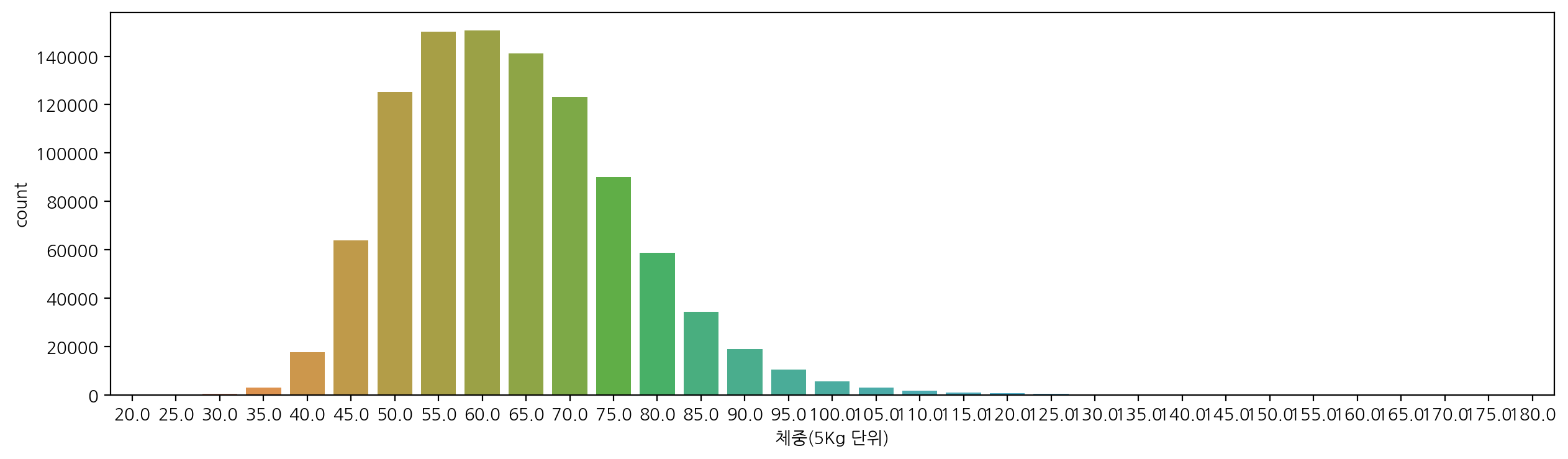
plt.figure(figsize=(15, 4))
sns.countplot(data=df, x="체중(5Kg 단위)", hue="성별코드")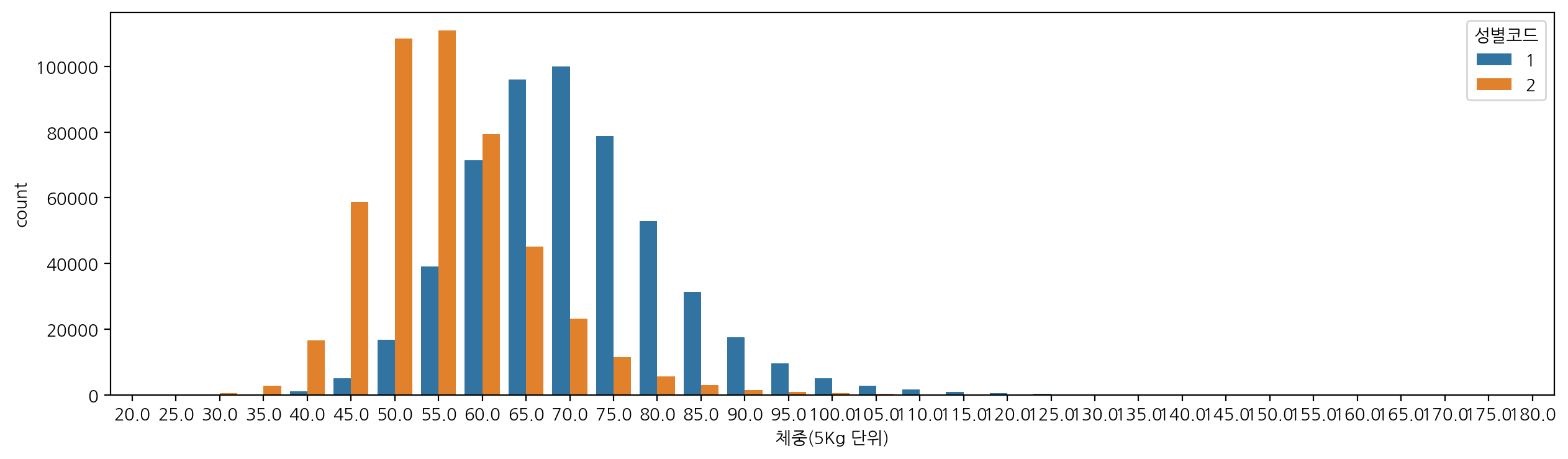
cf) 여기서 연령, 신장, 체중은 본래는 연속형 데이터이지만, 해당 데이터에서는 특정 범위로 묶어져 있으므로 연속형 데이터라기 보다는 범주형 데이터라고 볼 수 있다.
범주형 데이터에 대한 수치형 데이터 혹은 범주형 데이터를 막대그래프로 확인하고자 할 때 사용한다.
연령대코드별 수치형 데이터인 총콜레스테롤을 음주여부에 따라 확인해보자.
sns.barplot(data=df_sample, x="연령대코드(5세단위)", y="총콜레스테롤", hue="음주여부")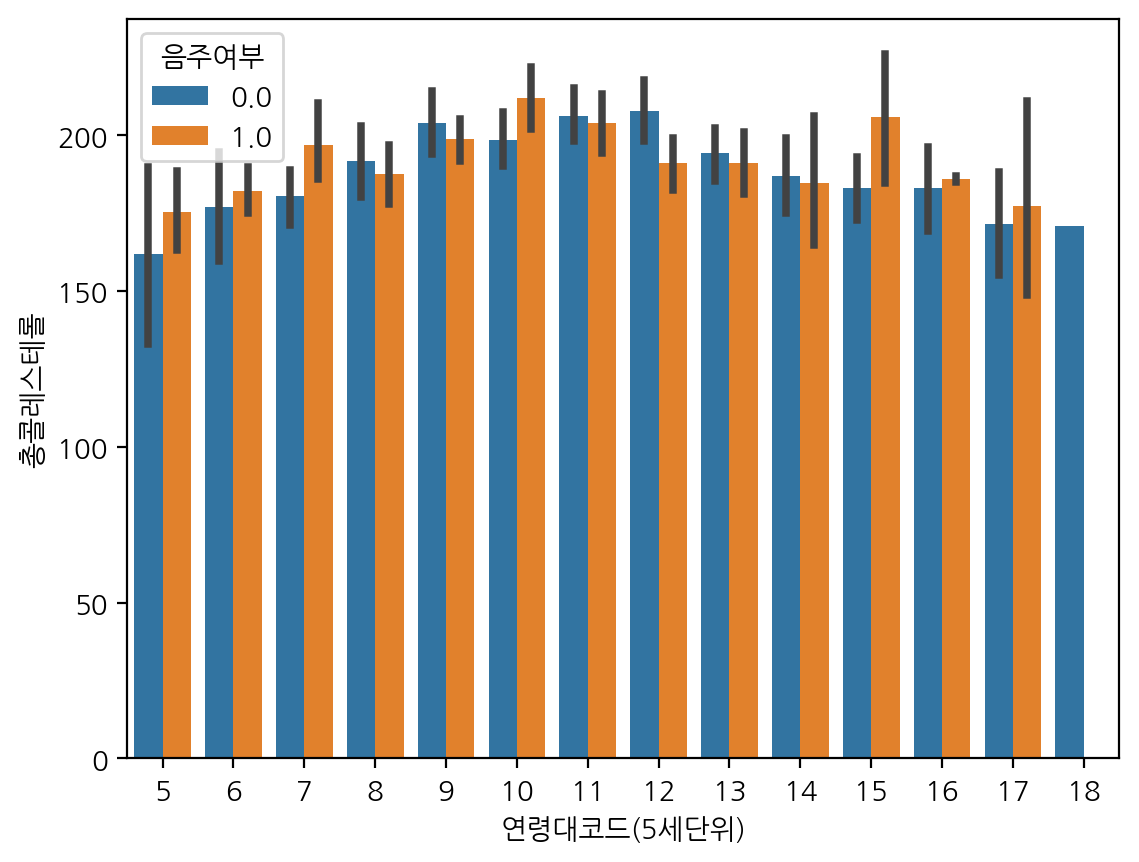
흡연상태에 따라서도 확인해보자.
plt.figure(figsize=(15, 4))
sns.barplot(data=df_sample, x="연령대코드(5세단위)", y="총콜레스테롤", hue="흡연상태")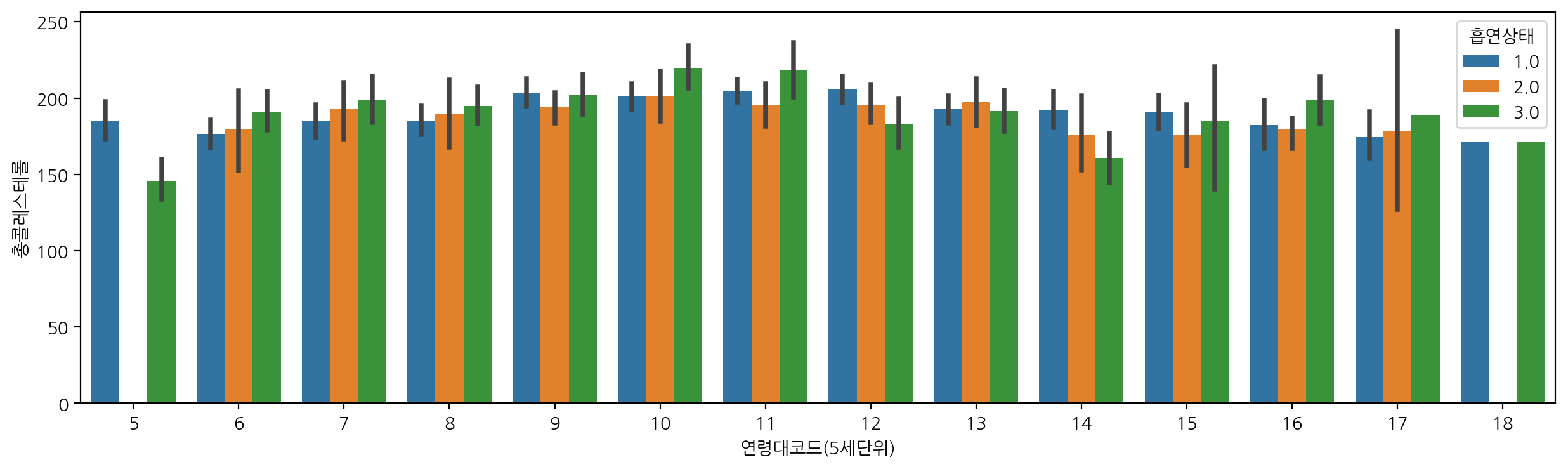
barplot에서 검은색 막대는 errorbar로 오차막대를 의미한다.
해당 오차막대의 범위는 '부트스트랩 신뢰구간'을 나타내고, 기본값은 95% 신뢰구간이다.
plot에 따라 'ci=값' 혹은 errorbar=('ci', 값)로 값을 조정할 수 있다.
(2023년도 기준, 특정 plot에 대해서는 경고 문구는 뜨지만 호환은 서로 가능한 상태)
다음은 연령대코드별 범주형 데이터인 체중을 성별코드와 음주여부에 따라 확인해보자.
# ci=None - 경고문구 확인
sns.barplot(data=df, x="연령대코드(5세단위)", y="체중(5Kg 단위)", hue="성별코드", ci=None)
# errorbar=None
sns.barplot(data=df, x="연령대코드(5세단위)", y="체중(5Kg 단위)", hue="성별코드", errorbar=None)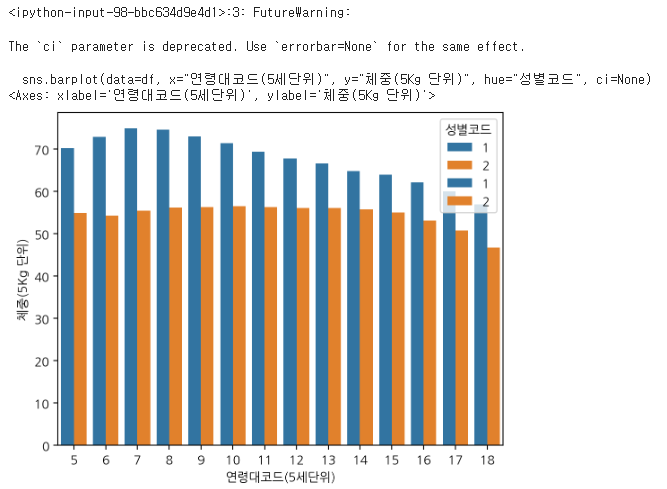
errorbar='sd'를 넣어 표준편차로 표현할 수 있다.
sns.barplot(data=df, x="연령대코드(5세단위)", y="체중(5Kg 단위)", hue="음주여부", errorbar='sd')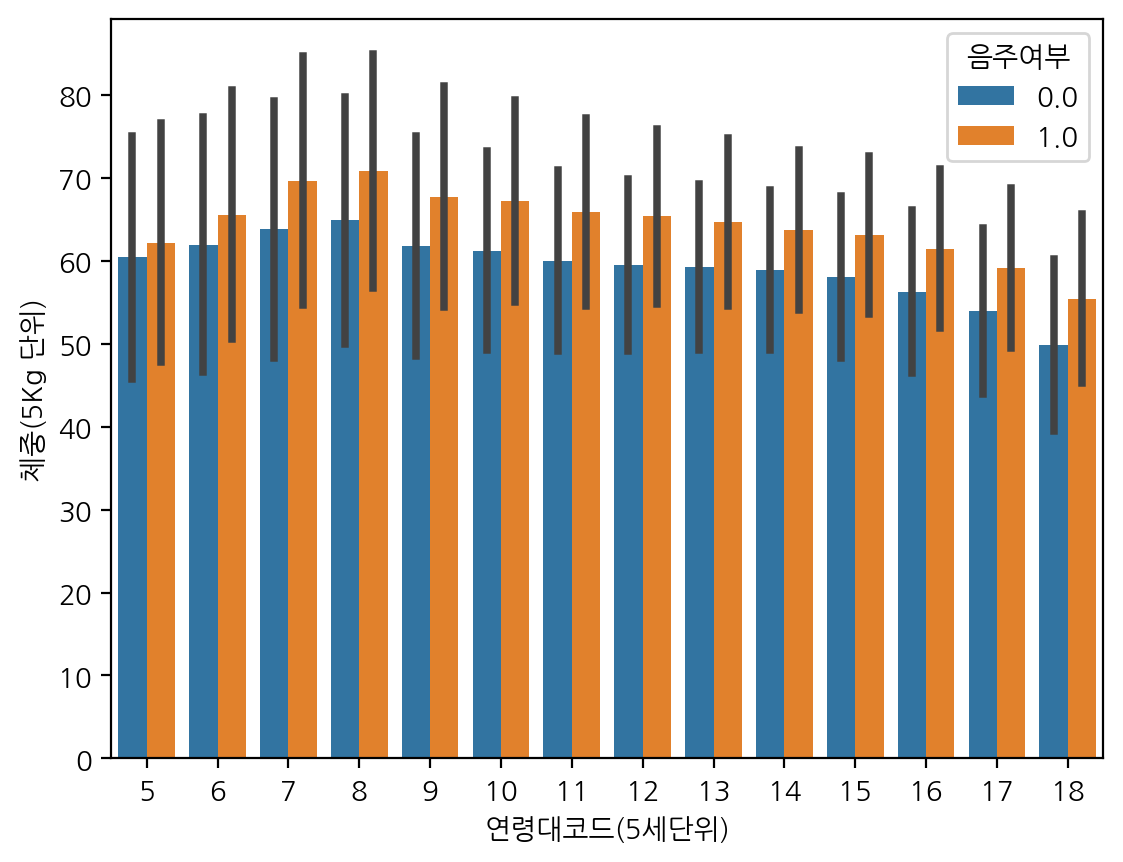
barplot과 달리 선으로 그래프를 그리는 방법이다. barplot과 동일하게 errorbar를 통해 오차막대 값을 조정할 수 있다.
연령대코드에 따른 체중을 성별코드에 따라 lineplot을 그려보자.
plt.figure(figsize=(15, 4))
sns.lineplot(data=df_sample, x="연령대코드(5세단위)", y="체중(5Kg 단위)", hue="성별코드", errorbar='sd')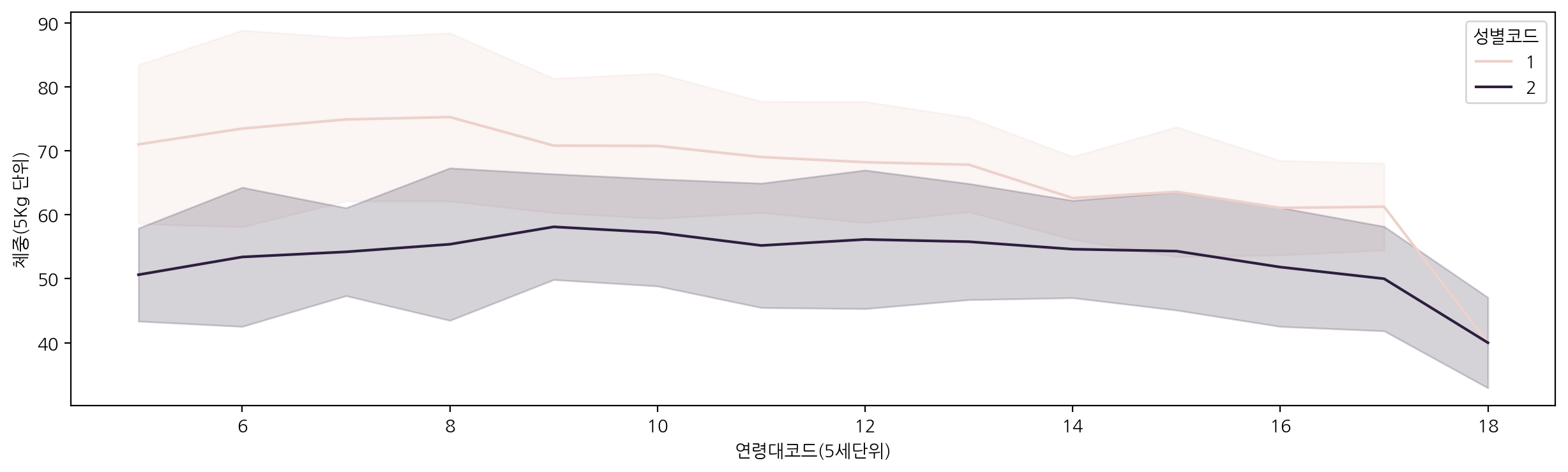
연령대코드에 따른 신장을 성별코드에 따라 lineplot을 그려보자.
plt.figure(figsize=(15, 4))
sns.lineplot(data=df_sample, x="연령대코드(5세단위)", y="신장(5Cm단위)", hue="성별코드", errorbar='sd')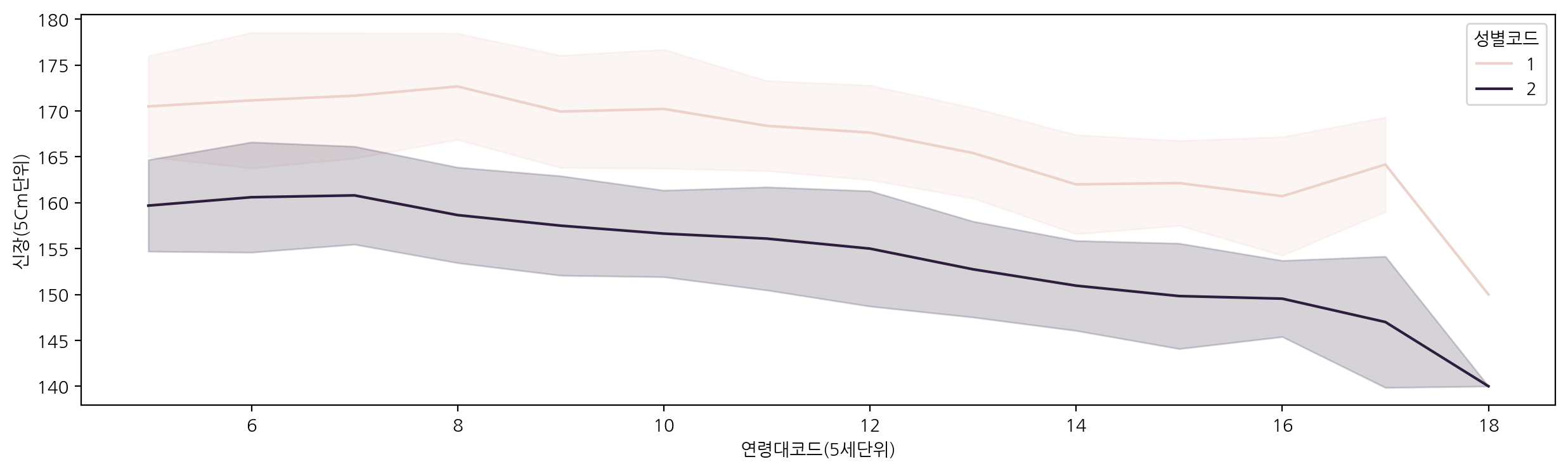
연령대코드에 따른 혈색소를 음주여부에 따라 lineplot을 그려보자.
plt.figure(figsize=(15, 4))
sns.lineplot(data=df, x="연령대코드(5세단위)", y="혈색소", hue="음주여부", errorbar=None)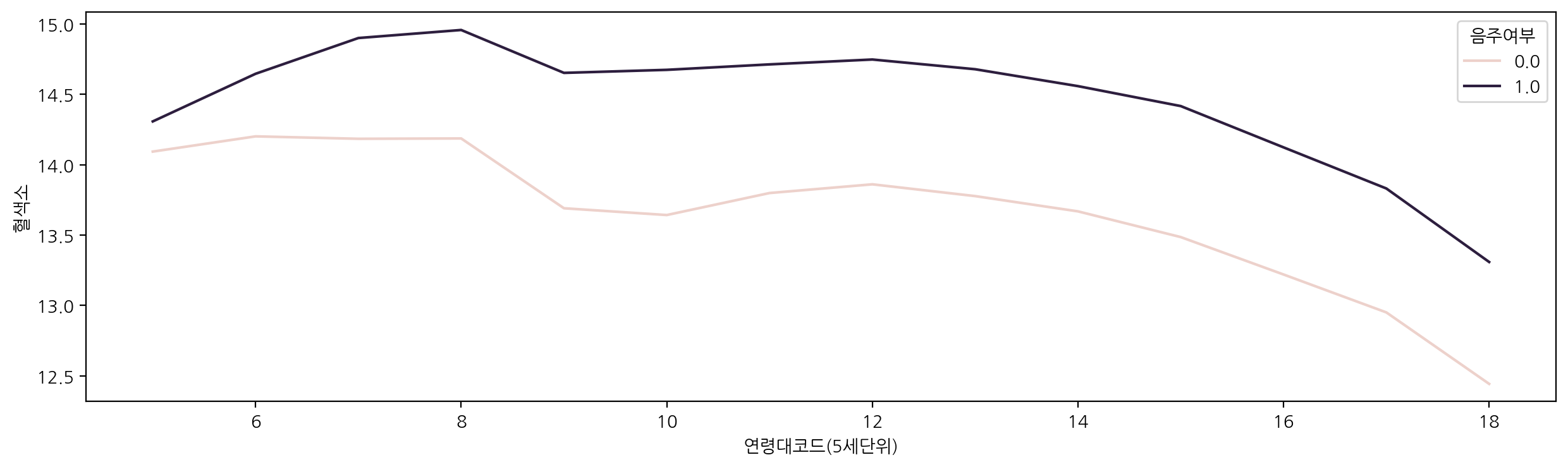
pointplot은 barplot과 lineplot과 비슷한데 점으로 그릴 수 있다.
연령대코드에 따른 신장을 성별코드에 따라 pointplot을 그려보자.
plt.figure(figsize=(15, 4))
sns.pointplot(data=df_sample, x="연령대코드(5세단위)", y="신장(5Cm단위)", hue="성별코드", errorbar='sd')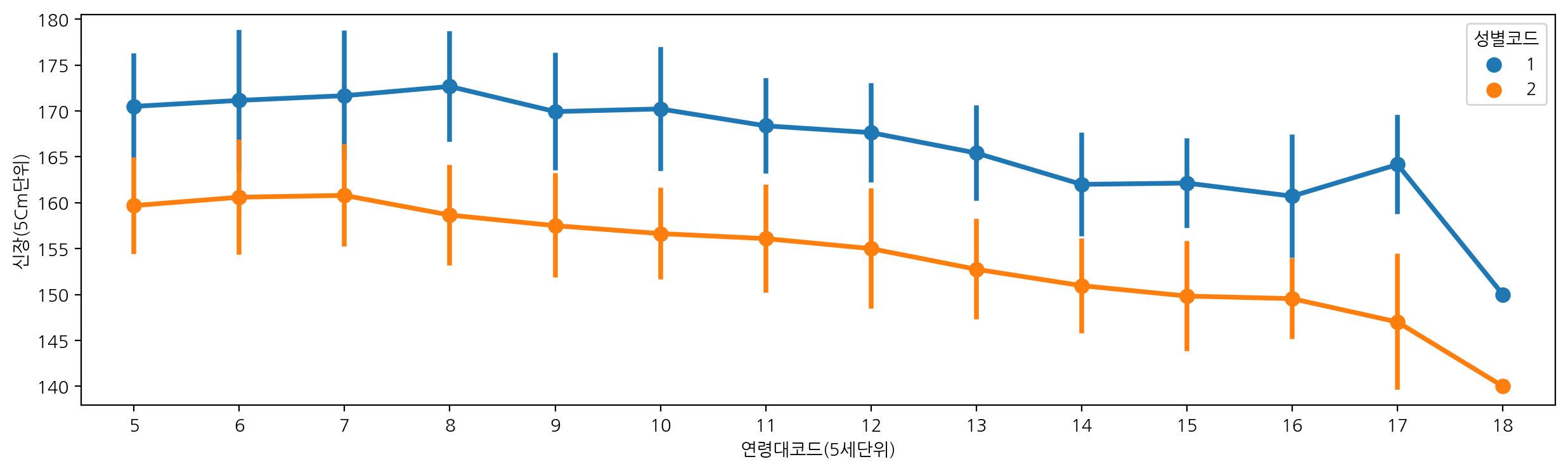
연령대코드에 따른 신장을 음주여부에 따라 barplot과 pointplot을 함께 그려보자.
plt.figure(figsize=(15, 4))
sns.barplot(data=df_sample, x="연령대코드(5세단위)", y="신장(5Cm단위)", hue="음주여부", errorbar='sd')
sns.pointplot(data=df_sample, x="연령대코드(5세단위)", y="신장(5Cm단위)", hue="음주여부", errorbar='sd')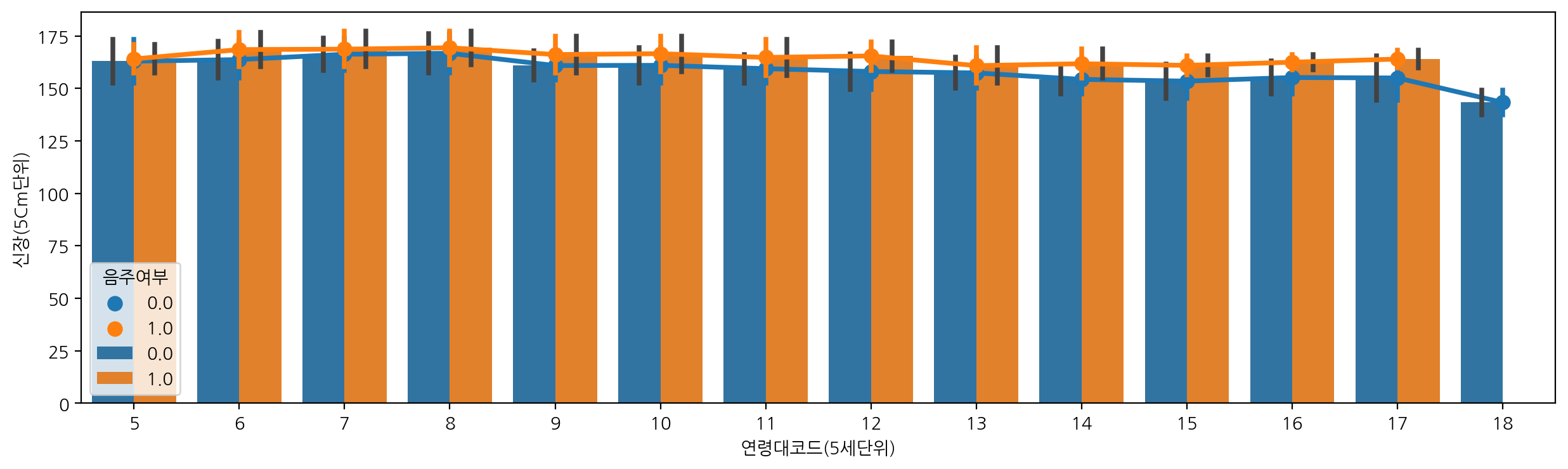
pandas.DataFrame.boxplot — pandas 2.1.3 documentation
Backend to use instead of the backend specified in the option plotting.backend. For instance, ‘matplotlib’. Alternatively, to specify the plotting.backend for the whole session, set pd.options.plotting.backend.
pandas.pydata.org
boxplot은 가공되지 않은 데이터를 그대로를 그린 것이 아니라, 데이터로 부터 얻어낸 통계량에 대한 5가지 요약 수치를 그린 것이다. 여기서 5가지 요약 수치란 최솟값, 제 1사분위수, 제 2사분위수(중앙값), 제 3사분위수, 최대값을 의미한다.
신장에 따른 체중을 성별코드에 따라 boxplot으로 그려보자.
plt.figure(figsize=(15, 4))
sns.boxplot(data=df, x="신장(5Cm단위)", y="체중(5Kg 단위)", hue="성별코드")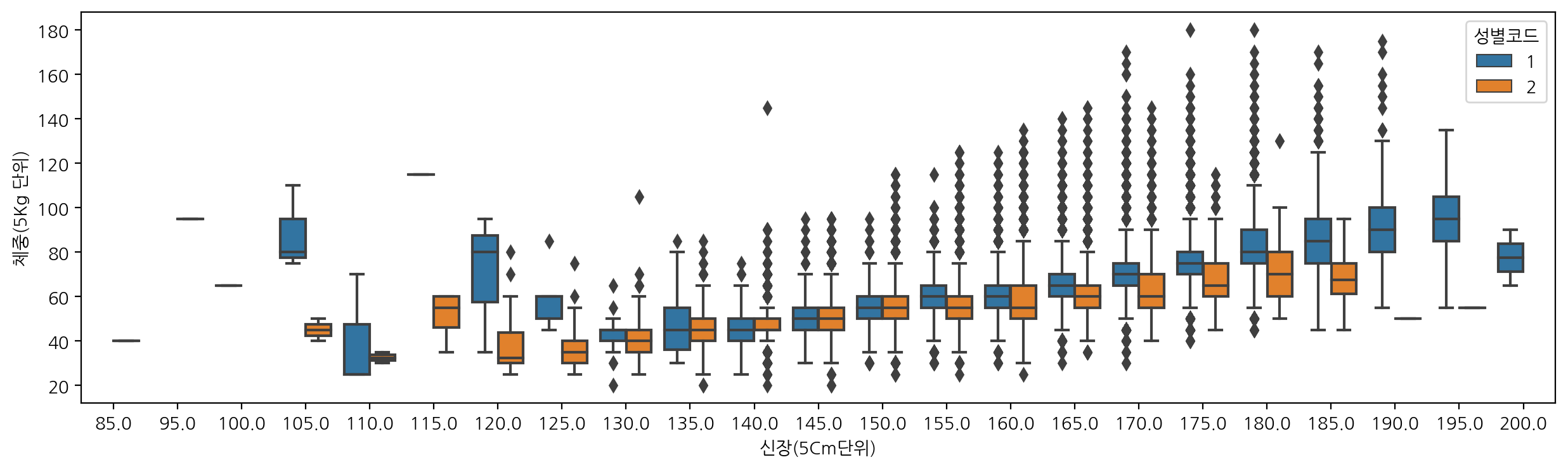
violinplot은 boxplot과 커널 밀도 추정 그래프(Kernel, Density Estimation, KDE)를 결합한 형태이다.
여기서 주요 요약 수치는 중앙값(중앙의 흰색점), KDE, boxplot, 바이올린 형태의 분포(주로 데이터가 어디에 분포되어 있는지 확인)가 있다.
신장에 따른 체중을 음주여부에 따라 violinplot으로 그려보자.
plt.figure(figsize=(15, 4))
sns.violinplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg 단위)", hue="음주여부")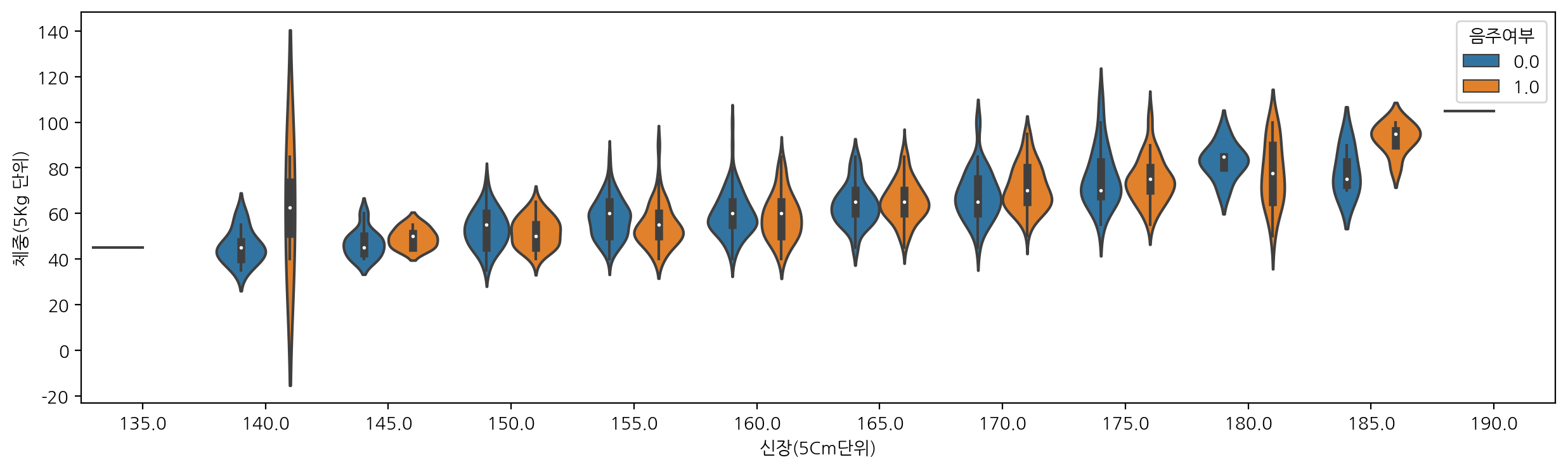
여기서 split 옵션을 사용해서 hue에서 지정한 컬럼을 대상으로 합쳐진 데이터에 대한 violinplot을 그릴 수 있다.
plt.figure(figsize=(15, 4))
sns.violinplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg 단위)", hue="음주여부", split=True)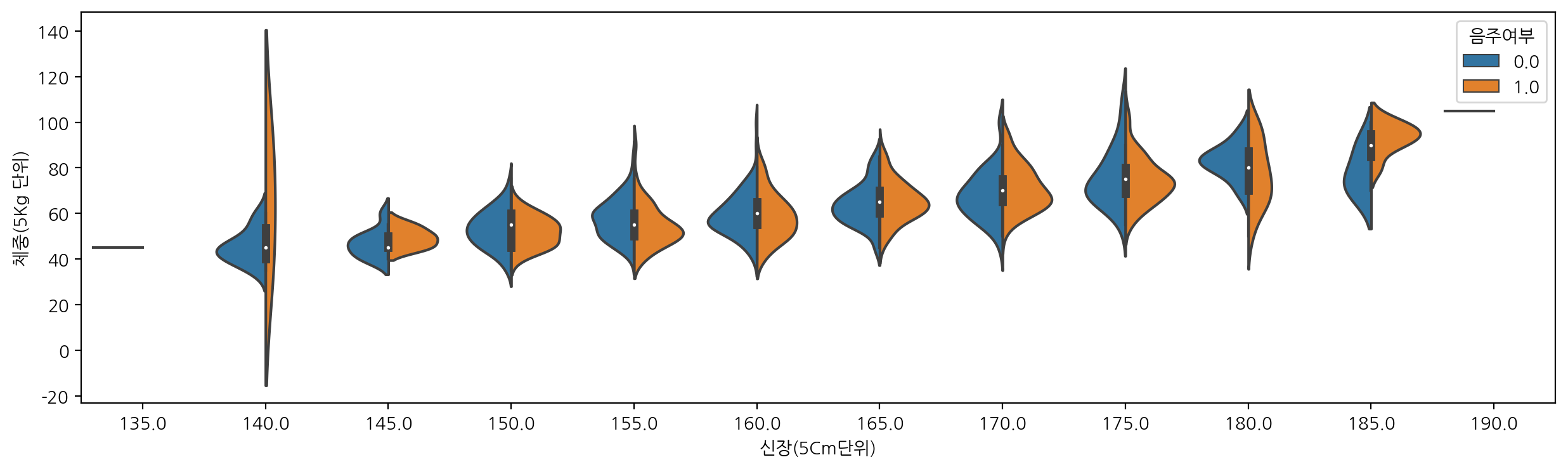
연령대코드에 따른 혈색소를 음주여부에 따라 violinplot으로 그려보자.
plt.figure(figsize=(15, 4))
sns.violinplot(data=df_sample, x="연령대코드(5세단위)", y="혈색소", hue="음주여부", split=True)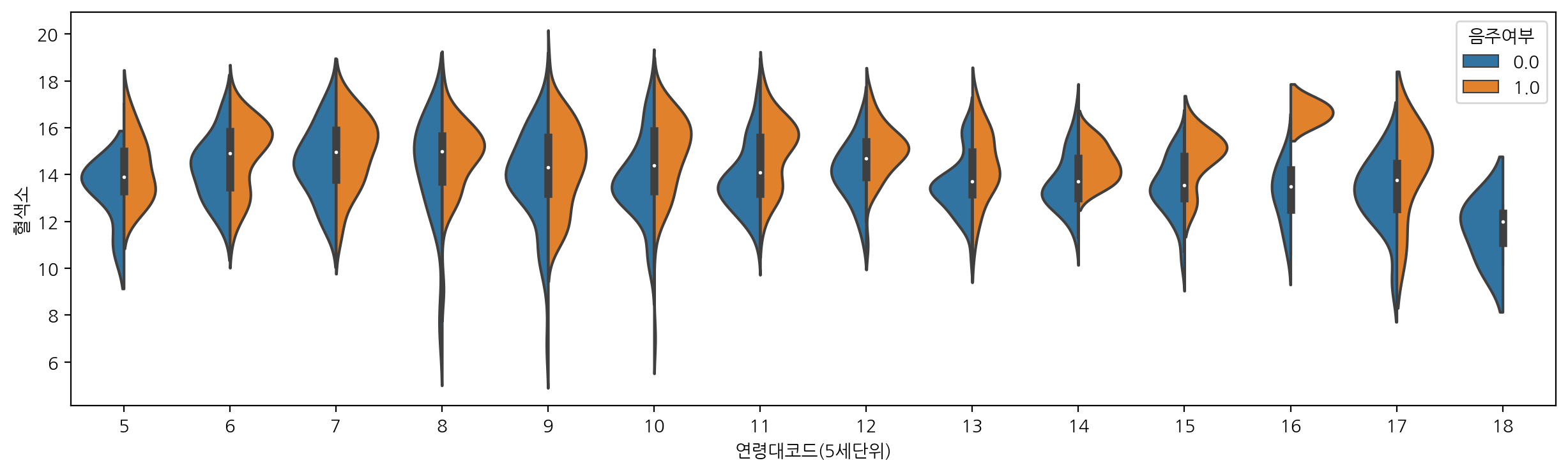
swarmplot은 범주형 데이터를 산점도로 시각화하고자 할 때 사용한다.
신장에 따른 체중을 음주여부에 따라 swarmplot으로 그려보자.
plt.figure(figsize=(15, 4))
sns.swarmplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg 단위)", hue="음주여부")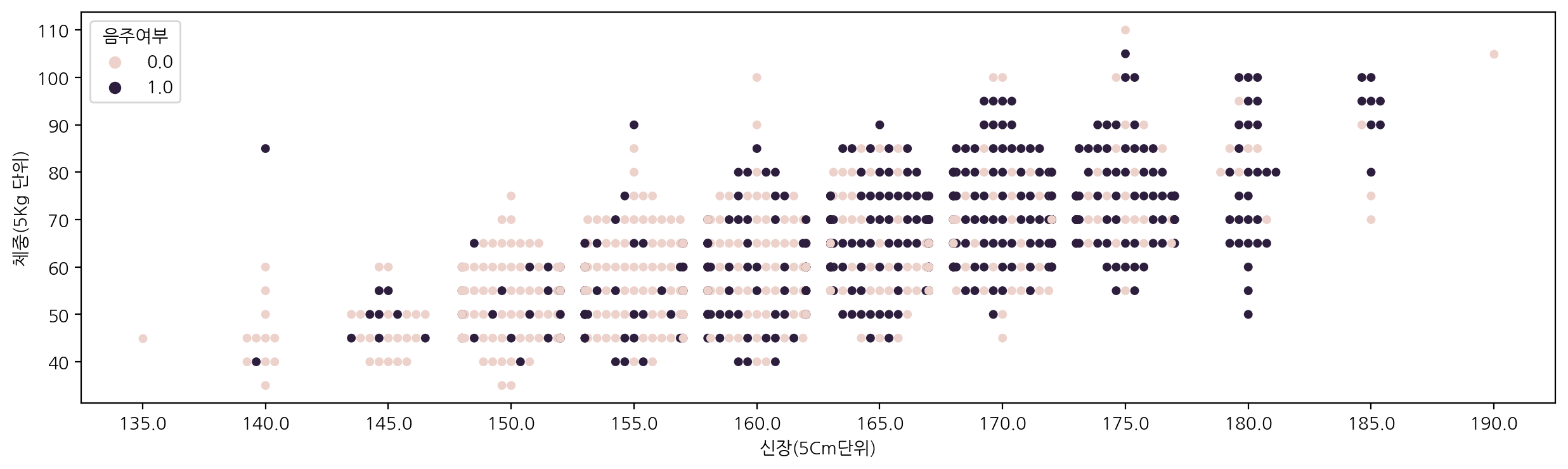
연령대코드에 따른 혈색소를 음주여부에 따라 swarmplot으로 그려보자.
plt.figure(figsize=(15, 4))
sns.swarmplot(data=df_sample, x="연령대코드(5세단위)", y="혈색소", hue="음주여부")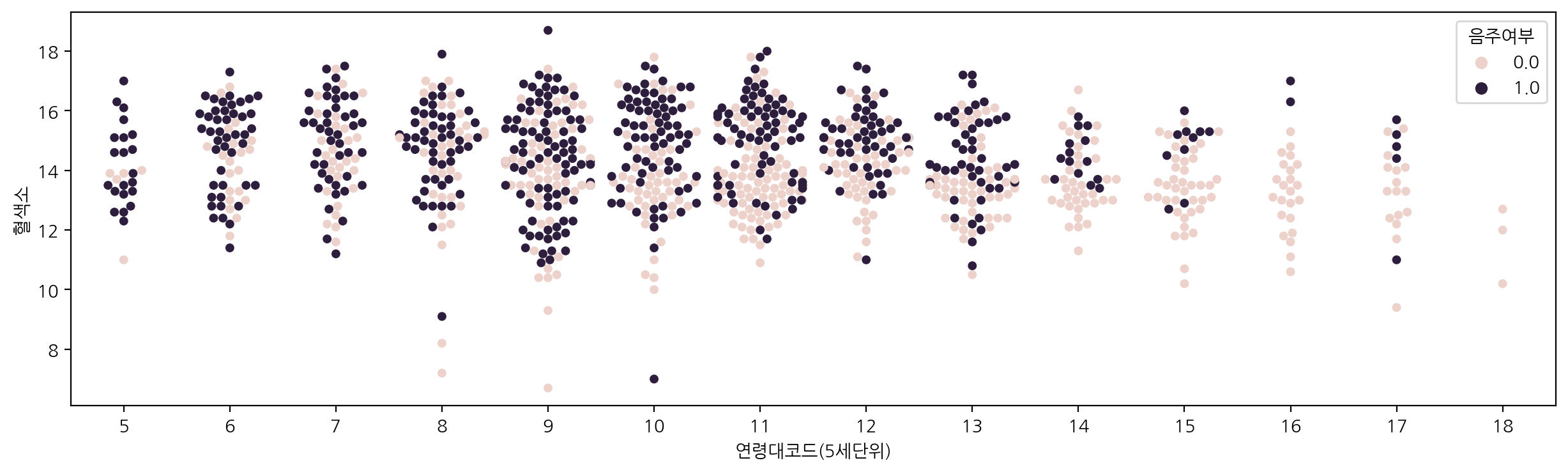
scatterplot은 수치형 데이터간의 상관관계를 볼 때 주로 사용한다.
점의 크기를 데이터의 수치에 따라 다르게 볼 수 있다.
"(혈청지오티)AST", "(혈청지오티)ALT" 을 그리고 음주여부에 따라 다른 색상으로 표현되게 하고, 체중에 따라 점의 크기를 다르게 scatterplot을 그려보자.
plt.figure(figsize=(8, 7))
sns.scatterplot(data=df_sample, x="(혈청지오티)AST", y="(혈청지오티)ALT",
hue="음주여부", size="체중(5Kg 단위)")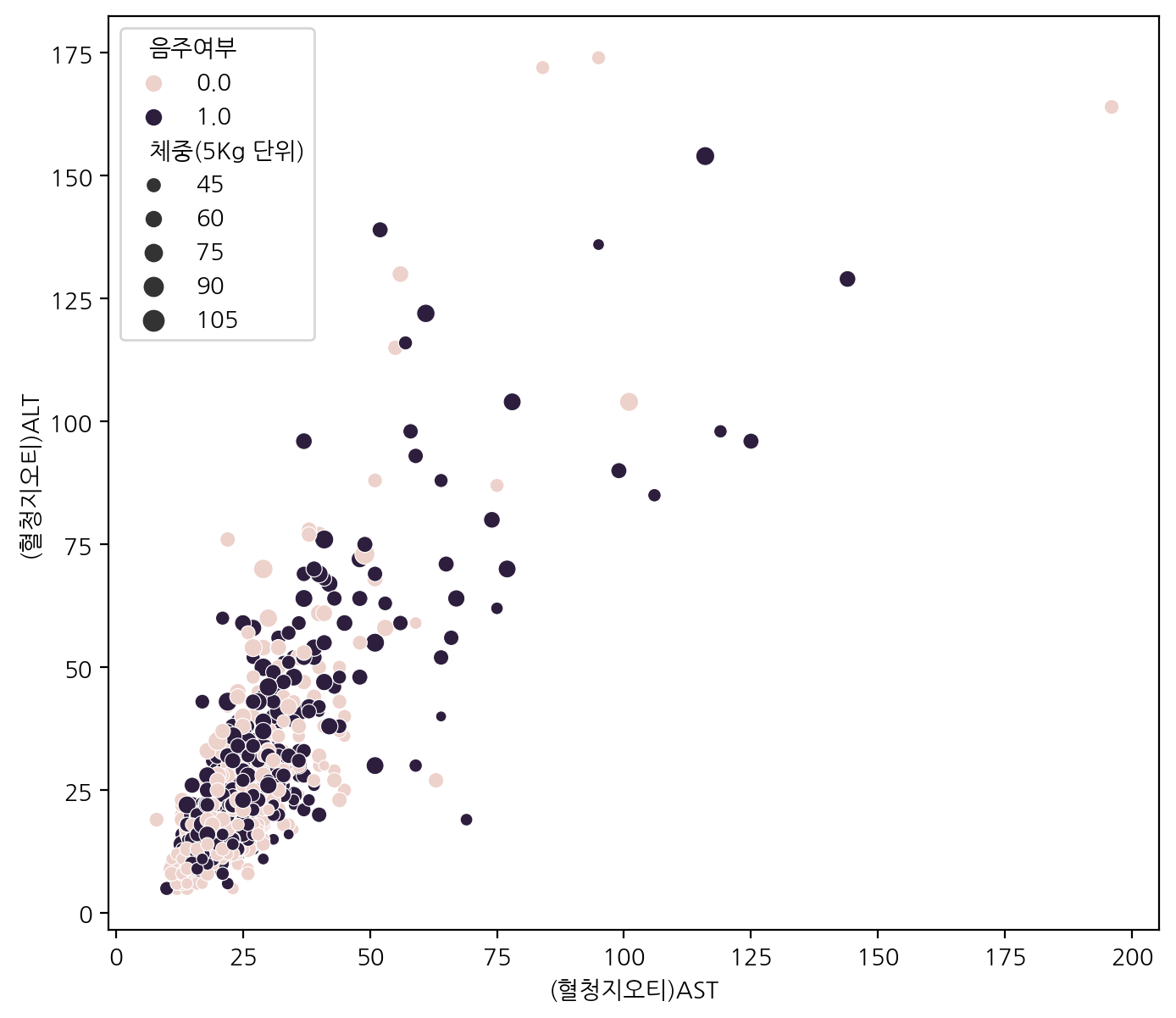
lmplot은 선형회귀 모델을 시각화하는데 사용된다. 데이터를 scatterplot으로 나타내고, 그 위에 선형회귀 모델의 적합한 선을 그려 변수 간의 상관관계를 확인할 수 있다.
신장에 따른 체중을 음주여부에 따라 표현하고, 성별코드로 열을 나눠서 lmplot으로 그려보자.
sns.lmplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg 단위)", hue="음주여부", col="성별코드")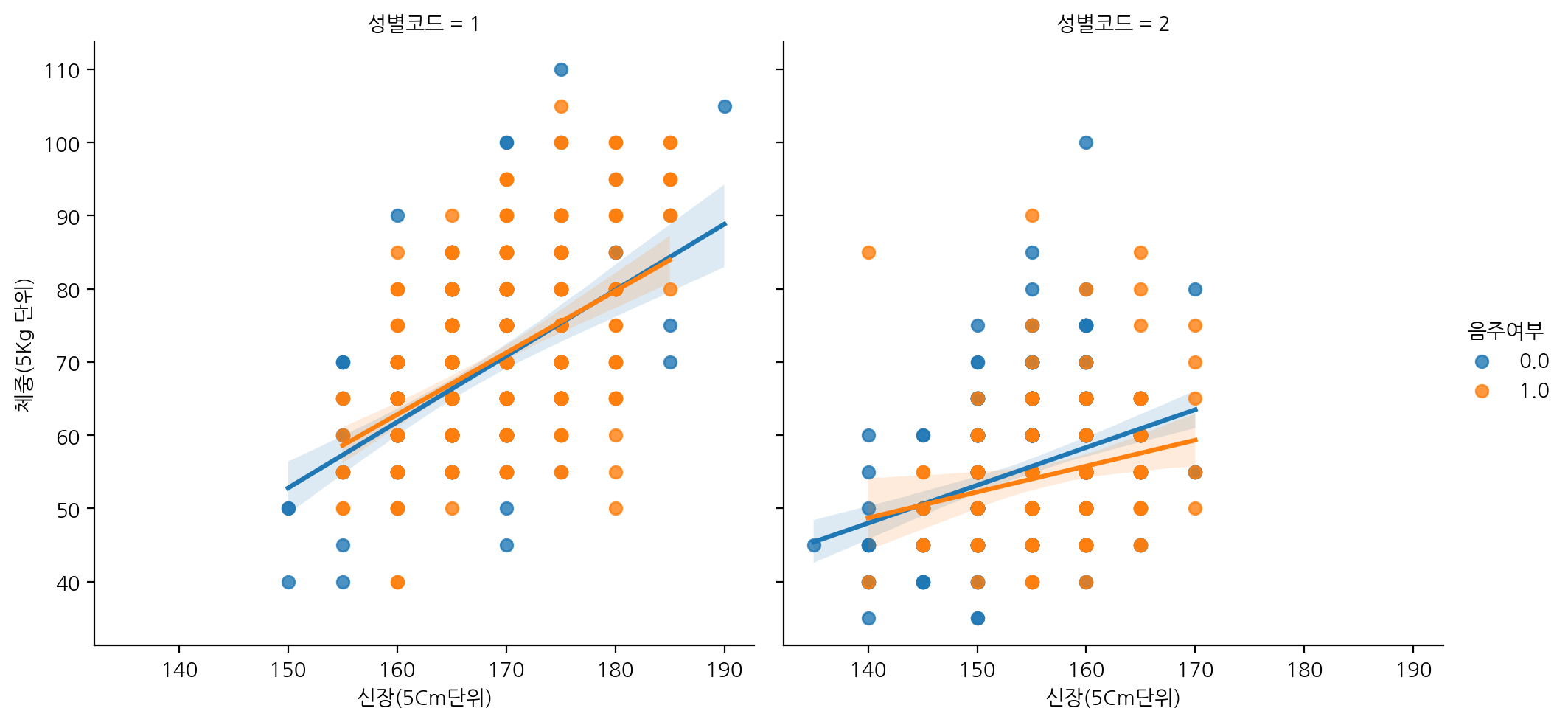
신장에 따른 체중을 성별코드에 따라 표현하고, 음주여부로 열을 나눠서 lmplot으로 그려보자.
sns.lmplot(data=df_sample, x="신장(5Cm단위)", y="체중(5Kg 단위)", hue="성별코드", col="음주여부")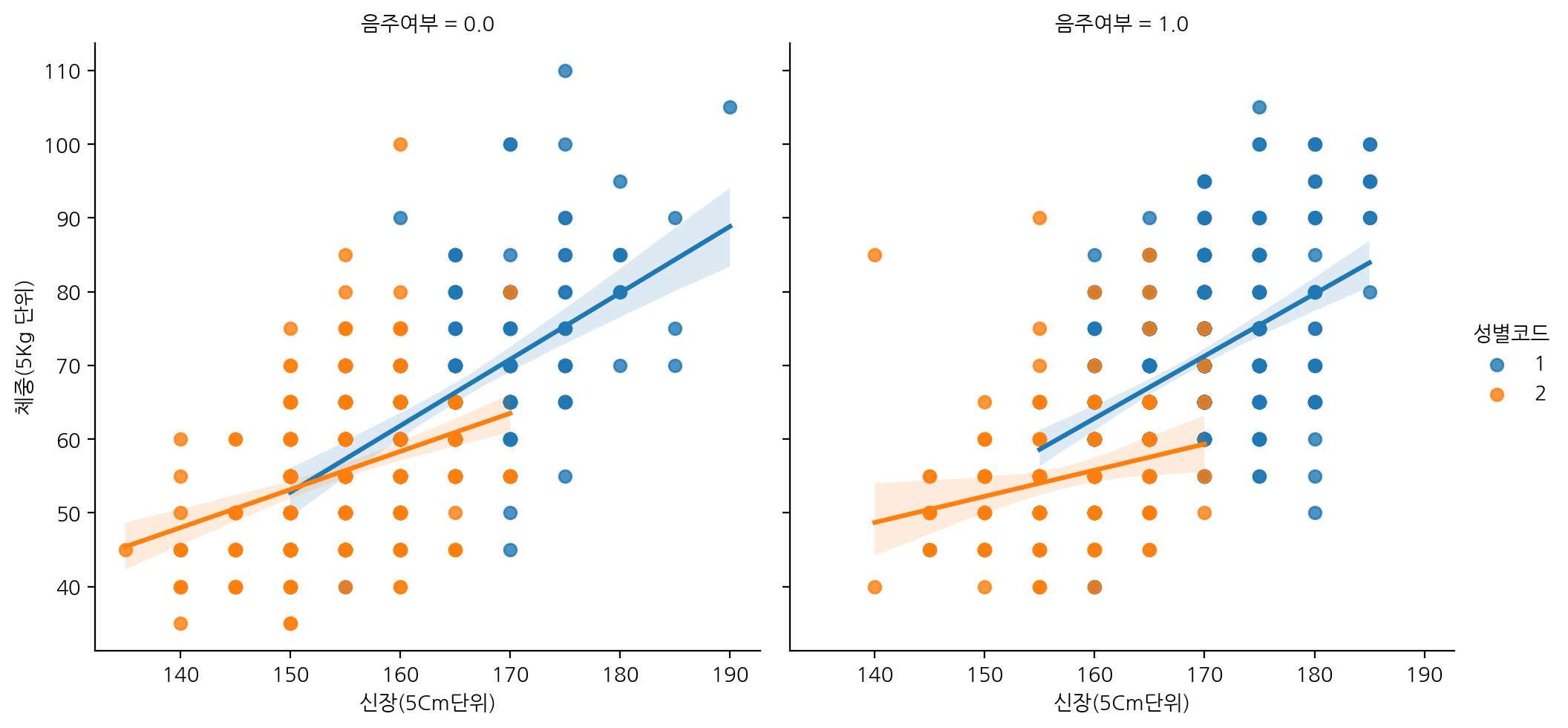
수축기혈압에 따른 이완기혈압을 음주여부에 따라 lmplot으로 그려보자.
sns.lmplot(data=df_sample, x="수축기혈압", y="이완기혈압", hue="음주여부")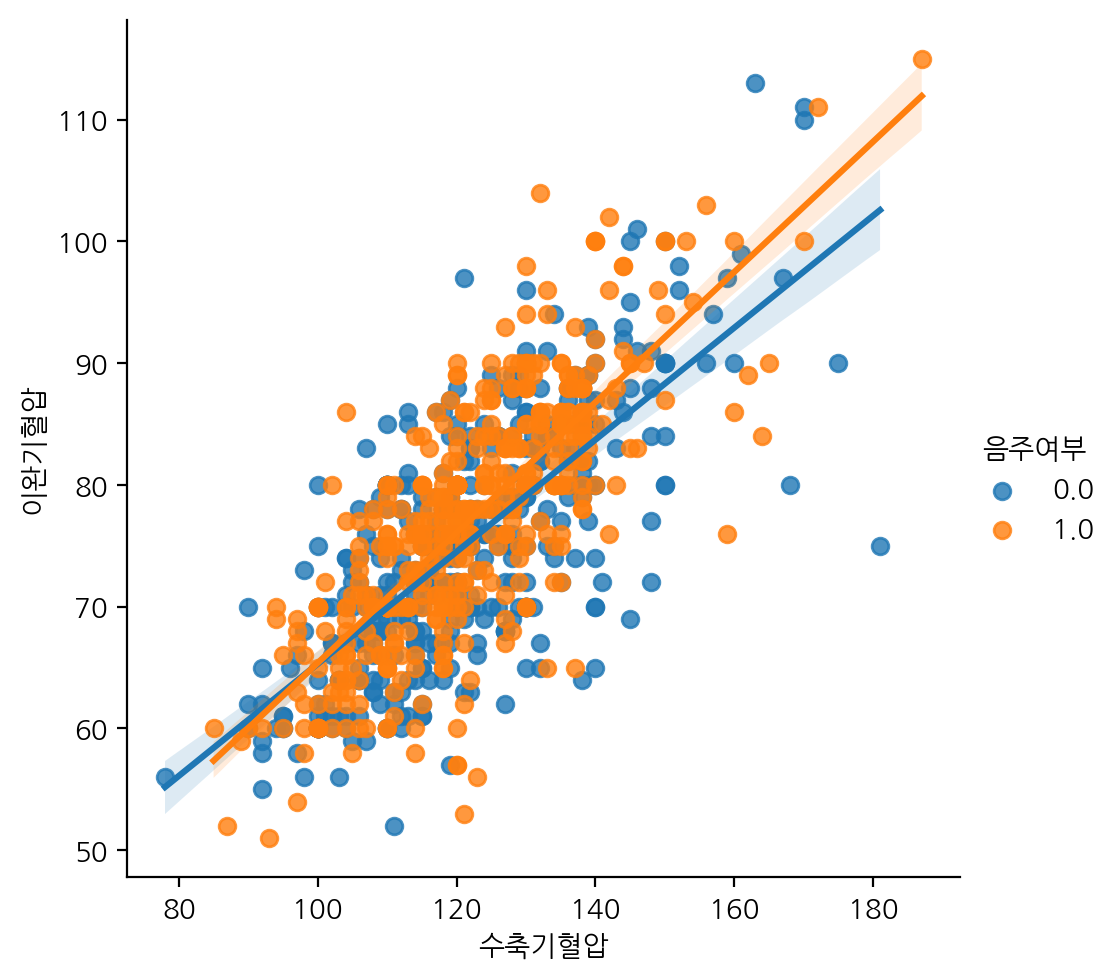
연령대코드에 따른 혈색소를 음주여부에 따라 표현하고, 성별코드로 열을 나눠서 lmplot으로 그려보자.
sns.lmplot(data=df_sample, x="연령대코드(5세단위)", y="혈색소", hue="음주여부", col="성별코드")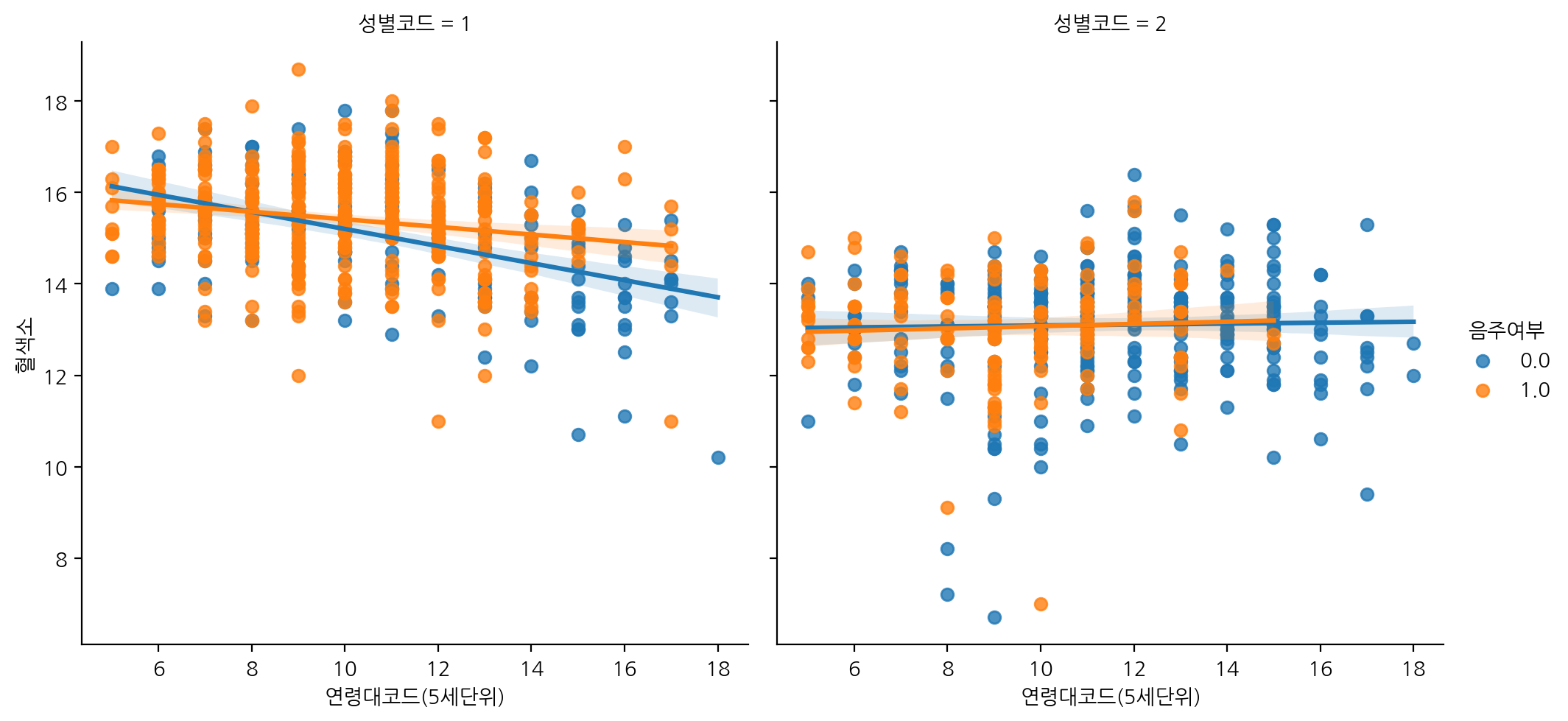
"(혈청지오티)AST", "(혈청지오티)ALT" 을 그리고 음주여부에 따라 다른 색상으로 lmplot으로 그려보자.
sns.lmplot(data=df_sample, x="(혈청지오티)AST", y="(혈청지오티)ALT", hue="음주여부", robust=True)
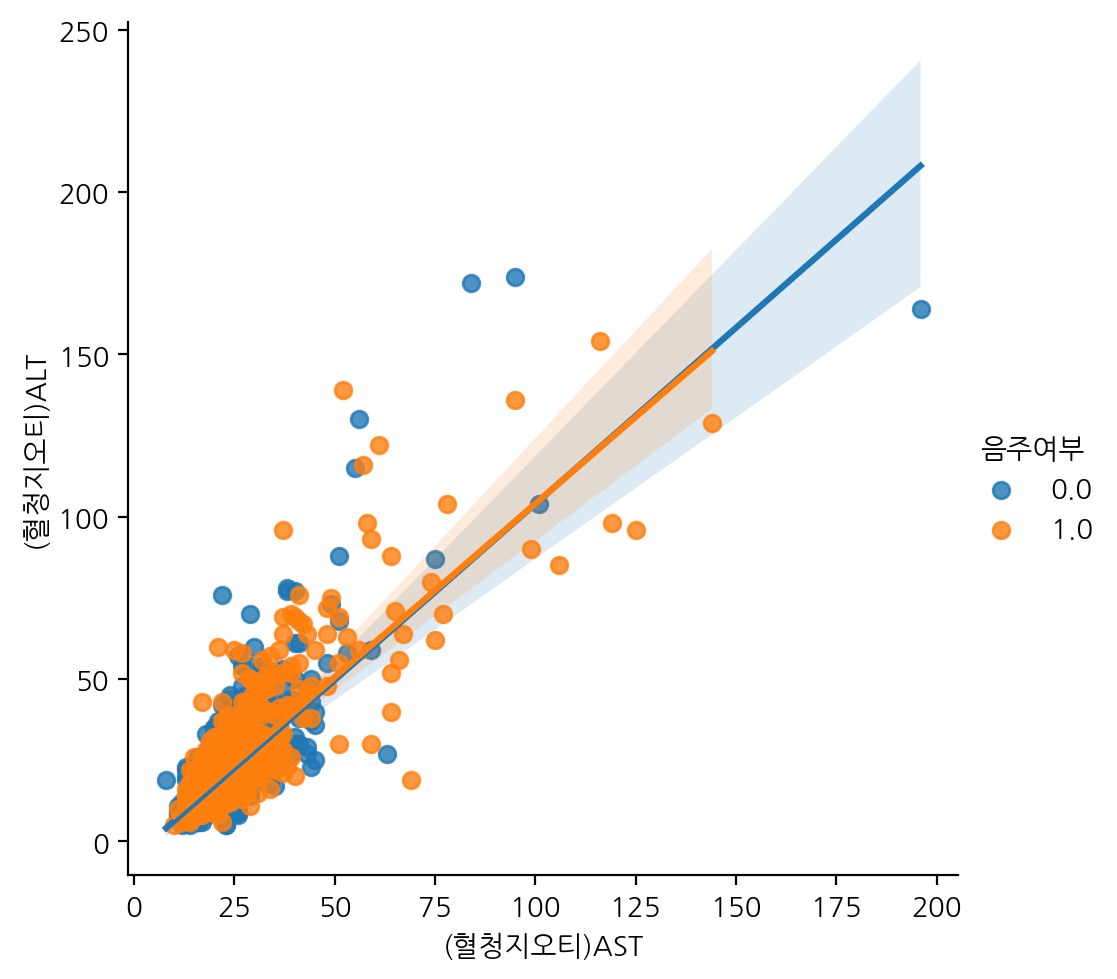
여기서 이상치가 있으면 회귀선이 달라지기도 하므로, 시각화를 통해 찾은 이상치만을 제거하여 따로 시각화해보자.
# 이상치 제거 데이터
df_ASLT = df_sample[(df_sample["(혈청지오티)AST"] < 400) & (df_sample["(혈청지오티)ALT"] < 400)]
sns.lmplot(data=df_ASLT, x="(혈청지오티)AST", y="(혈청지오티)ALT", hue="음주여부", ci=None)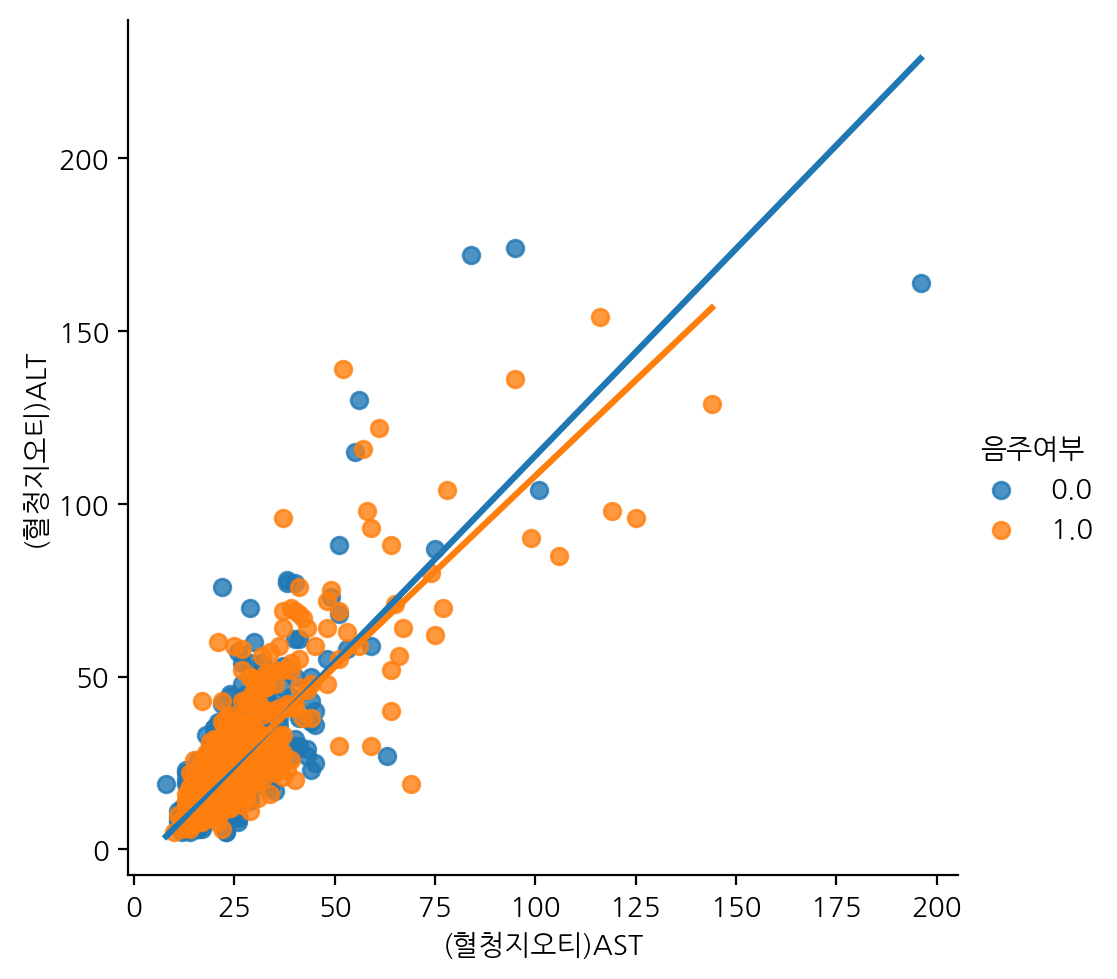
# 이상치 데이터
df_ASLT_high = df[(df["(혈청지오티)AST"] > 400) | (df["(혈청지오티)ALT"] > 400)]
sns.lmplot(data=df_ASLT_high, x="(혈청지오티)AST", y="(혈청지오티)ALT", hue="음주여부", ci=None)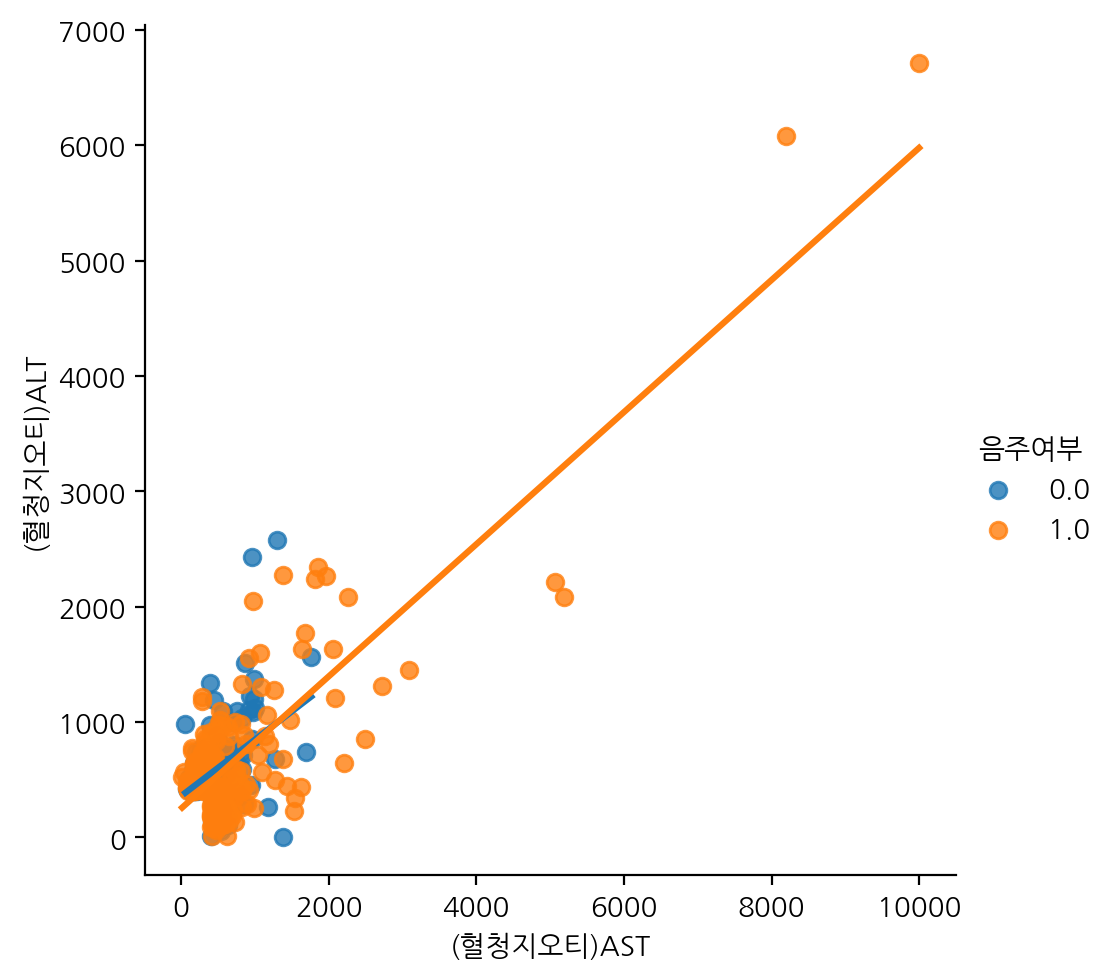
distplot은 히스토그램과 커널 밀도 추정 그래프(Kernel Density Estimation, KDE)를 결합하여 데이터의 분포를 확인할 수 있게 해준다. (향후 seaborn v0.14.0. 에서는 distplot이 사라지고, .displot이나 histplot으로 사용하는 것을 권장한다고 함)

여기서 총콜레스테롤에서 결측치를 제거한 값에 대해 distplot을 그려보자.
sns.distplot(df.loc[df["총콜레스테롤"].notnull() & (df["음주여부"] == 1), "총콜레스테롤"])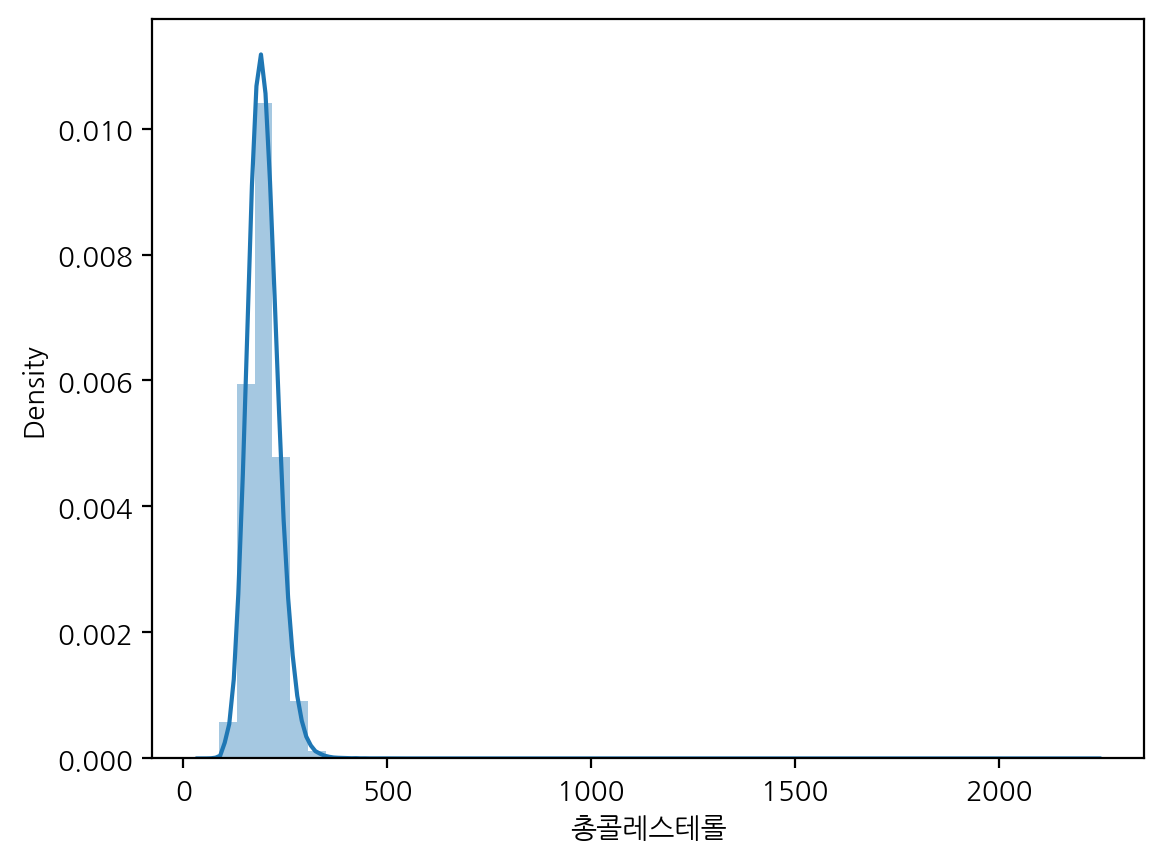
sns.distplot(df.loc[df["총콜레스테롤"].notnull() & (df["음주여부"] == 0), "총콜레스테롤"])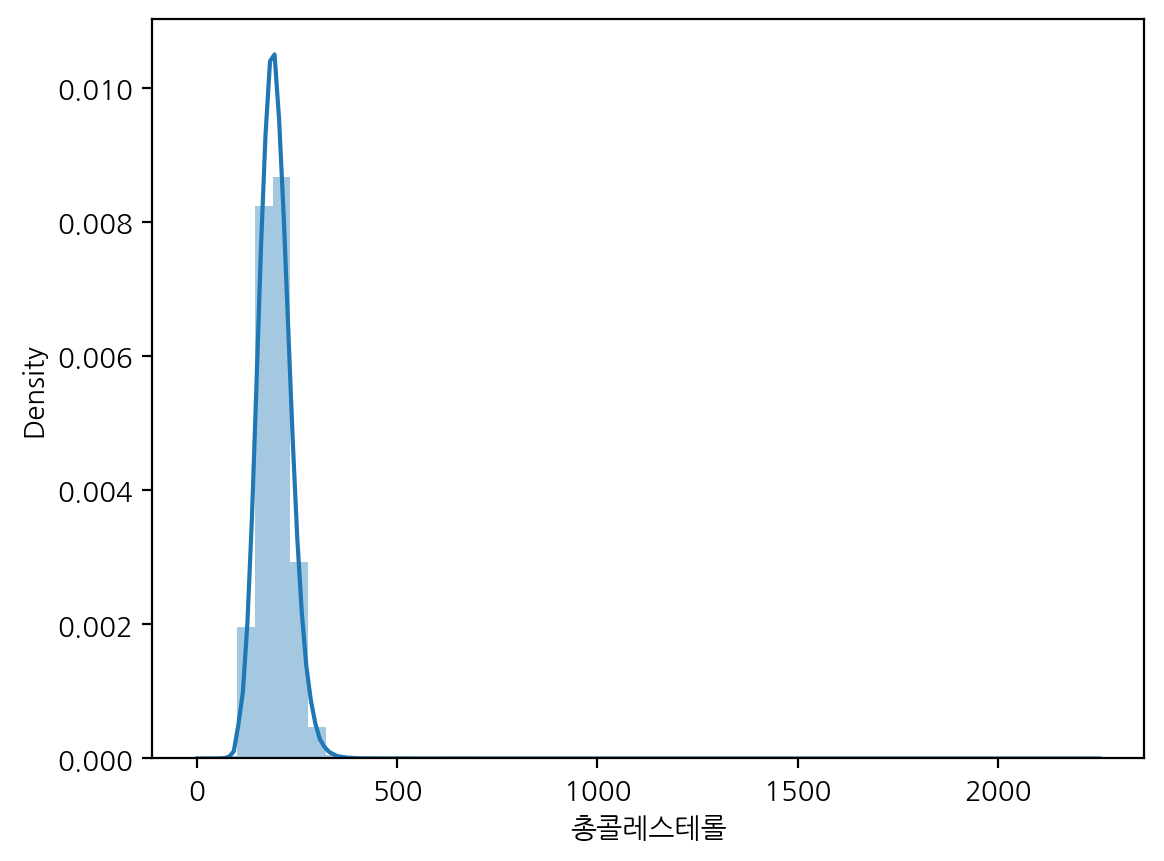
음주여부 값에 대한 총콜레스테롤을 하나의 그래프로 distplot으로 그려보자.
plt.axvline(df_sample["총콜레스테롤"].mean(), linestyle=":")
plt.axvline(df_sample["총콜레스테롤"].median(), linestyle="--")
sns.distplot(df_sample.loc[df_sample["총콜레스테롤"].notnull() & (df["음주여부"] == 1), "총콜레스테롤"], label="음주 중")
sns.distplot(df_sample.loc[df_sample["총콜레스테롤"].notnull() & (df["음주여부"] == 0), "총콜레스테롤"], label="음주 안 함")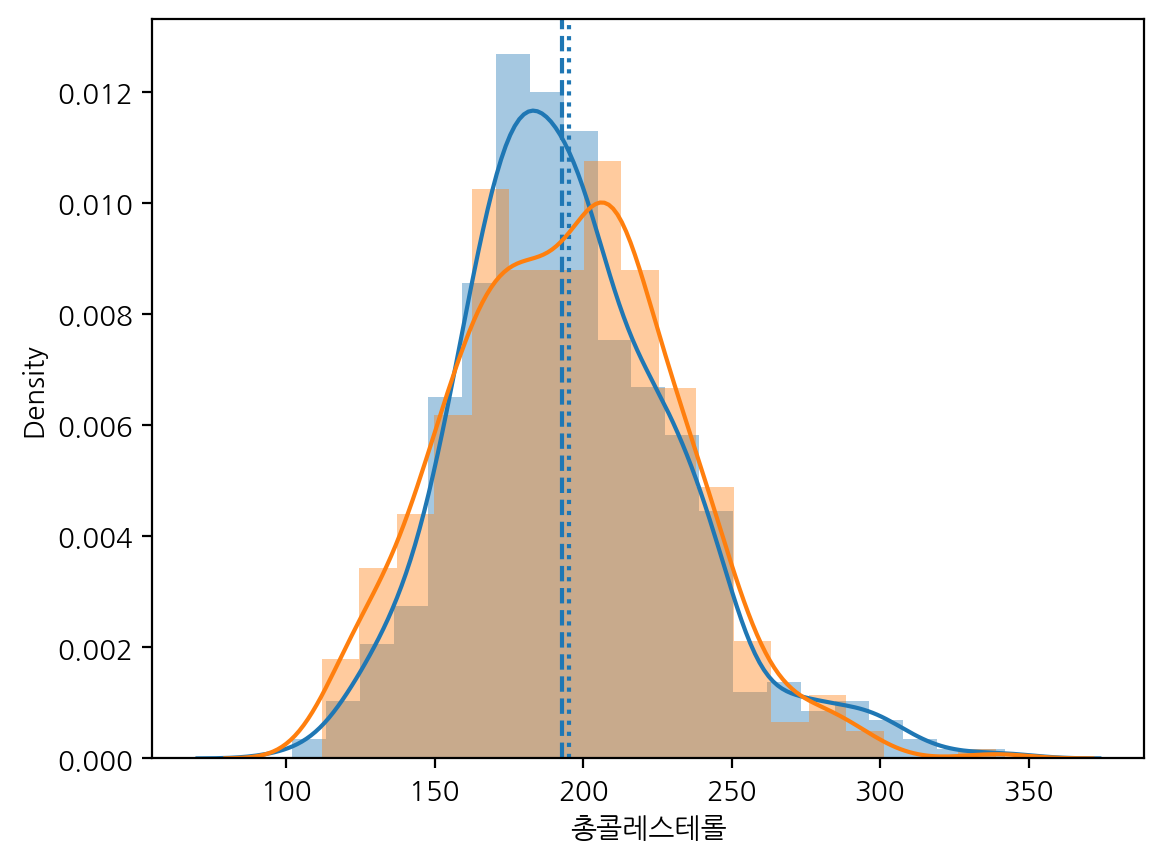
kdeplot은 커널 밀도 추정 그래프(Kernel Density Estimation, KDE)을 생성하여 데이터 분포를 확인할 수 있게 해준다.
kdeplot은 히스토그램의 단점을 보완하면서도 데이터의 분포를 더 부드럽게 표현할 수 있다.
음주여부 값에 대한 총콜레스테롤을 하나의 그래프로 kdeplot으로 그려보자.
plt.axvline(df_sample["총콜레스테롤"].mean(), linestyle=":")
plt.axvline(df_sample["총콜레스테롤"].median(), linestyle="--")
sns.kdeplot(df_sample.loc[df_sample["총콜레스테롤"].notnull() & (df["음주여부"] == 1), "총콜레스테롤"], label="음주 중")
sns.kdeplot(df_sample.loc[df_sample["총콜레스테롤"].notnull() & (df["음주여부"] == 0), "총콜레스테롤"], label="음주 안 함")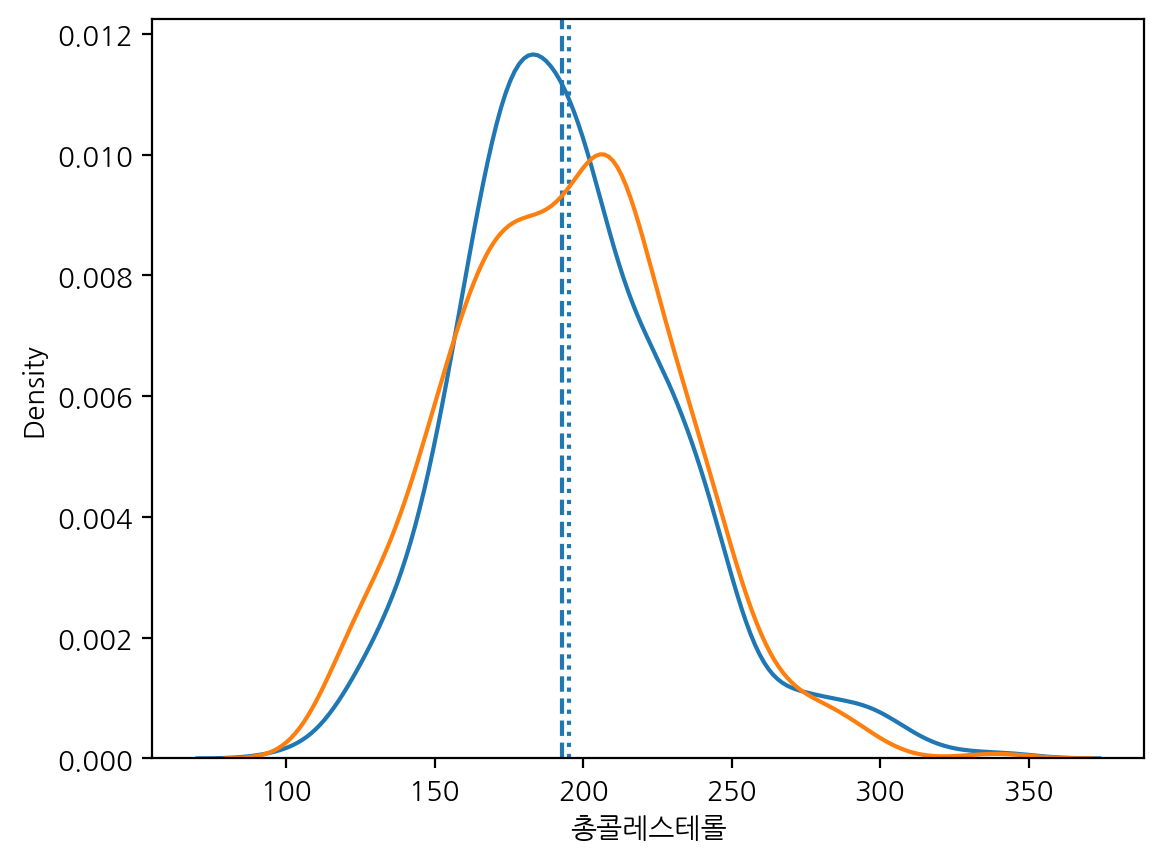
감마지피티 값에 따라 음주여부를 kdeplot으로 그려보자.
s_1 = df_sample.loc[df_sample["음주여부"] == 1, "감마지티피"]
s_0 = df_sample.loc[df_sample["음주여부"] == 0, "감마지티피"]
sns.kdeplot(s_1, label="음주 중")
sns.kdeplot(s_0, label="음주 안 함")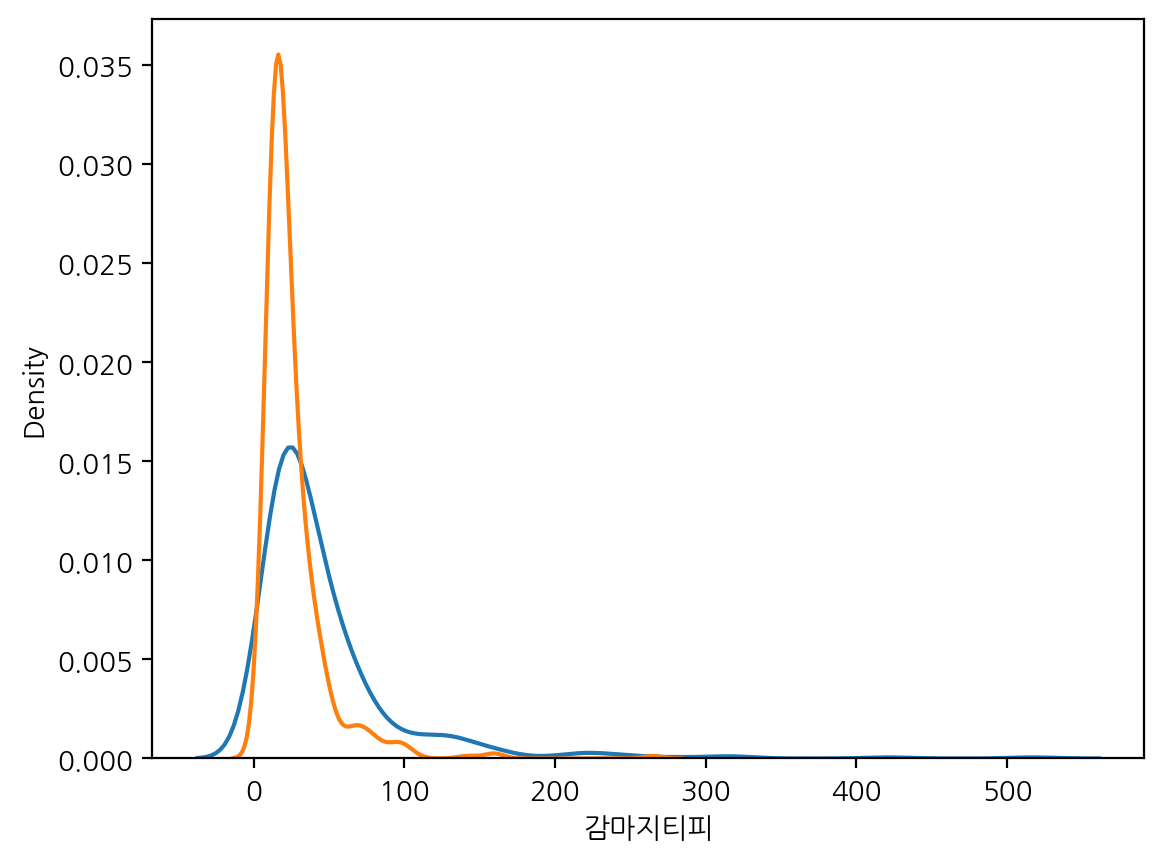
상관 분석 - 위키백과, 우리 모두의 백과사전
위키백과, 우리 모두의 백과사전. 상관 분석(相關 分析, 영어: correlation analysis, dependence analysis)은 확률론과 통계학에서 두 변수 간에 어떤 선형적 관계를 갖고 있는 지를 분석하는 방법이다. 두
ko.wikipedia.org
corr 함수를 통해서 컬럼간의 상관계수를 구할 수 있다.
상관계수(r)는 일반적으로 아래과 같이 해석한다.
| r이 -1.0과 -0.7 사이이면, 강한 음적 선형관계, r이 -0.7과 -0.3 사이이면, 뚜렷한 음적 선형관계, r이 -0.3과 -0.1 사이이면, 약한 음적 선형관계, r이 -0.1과 +0.1 사이이면, 거의 무시될 수 있는 선형관계, r이 +0.1과 +0.3 사이이면, 약한 양적 선형관계, r이 +0.3과 +0.7 사이이면, 뚜렷한 양적 선형관계, r이 +0.7과 +1.0 사이이면, 강한 양적 선형관계 |
# 상관계수에 사용할 컬럼들
columns = ['연령대코드(5세단위)', '체중(5Kg 단위)', '신장(5Cm단위)', '허리둘레',
'시력(좌)', '시력(우)', '청력(좌)', '청력(우)',
'수축기혈압', '이완기혈압', '식전혈당(공복혈당)',
'총콜레스테롤', '트리글리세라이드', 'HDL콜레스테롤', 'LDL콜레스테롤', '혈색소', '요단백',
'혈청크레아티닌', '(혈청지오티)AST', '(혈청지오티)ALT', '감마지티피', '흡연상태', '음주여부']
df_small = df_sample[columns]
df_corr = df_small.corr()
df_corr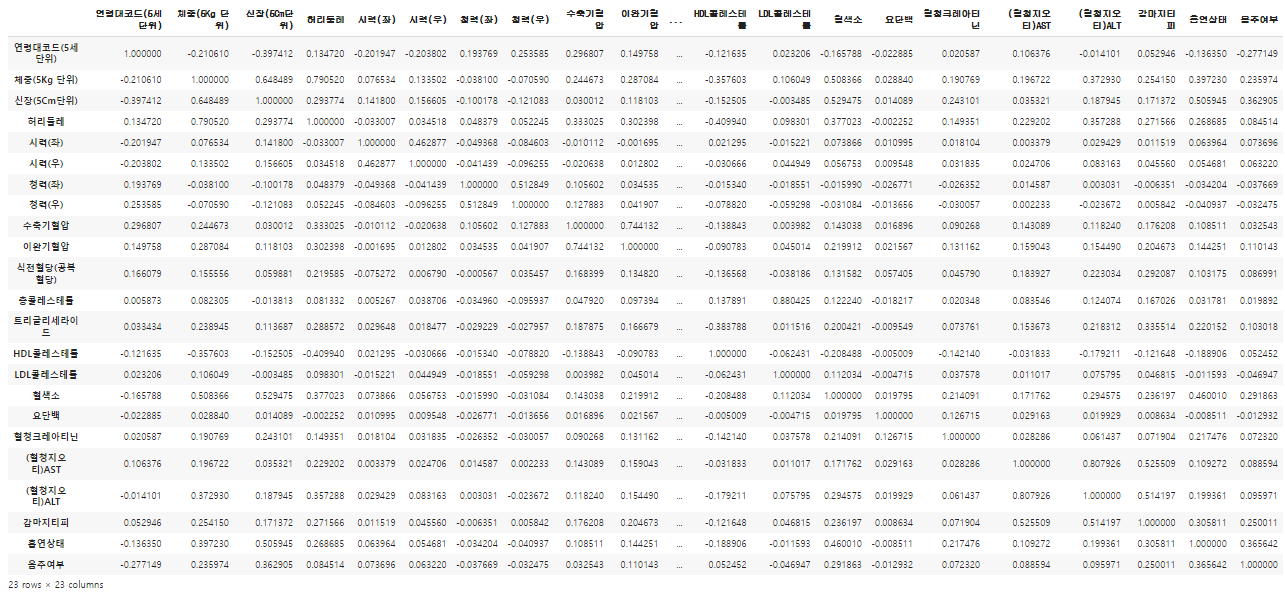
음주여부에 따른 상관계수가 특정 수치 이상인 데이터를 살펴보자.
df_corr.loc[df_corr["음주여부"] > 0.25,"음주여부"]
혈색소에 따른 상관계수가 높은 상위 7개의 데이터를 살펴보자.
df_corr["혈색소"].sort_values(ascending=False).head(7)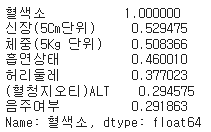
감마지피티에 따른 상관계수가 높은 상위 7개의 데이터를 살펴보자.
df_corr["감마지티피"].sort_values(ascending=False).head(7)
위에서 구한 상관계수를 heatmap으로 표현해보자.
annot=True 옵션을 통해 heatmap 위에 값을 같이 출력할 수 있고, fmt을 통해 출력값의 포맷을 지정할 수 있다.
또한, cmap으로 heatmap의 colormap을 지정해줄 수 있다.
plt.figure(figsize=(20, 7))
sns.heatmap(df_corr, annot=True, fmt=".2f", cmap="coolwarm")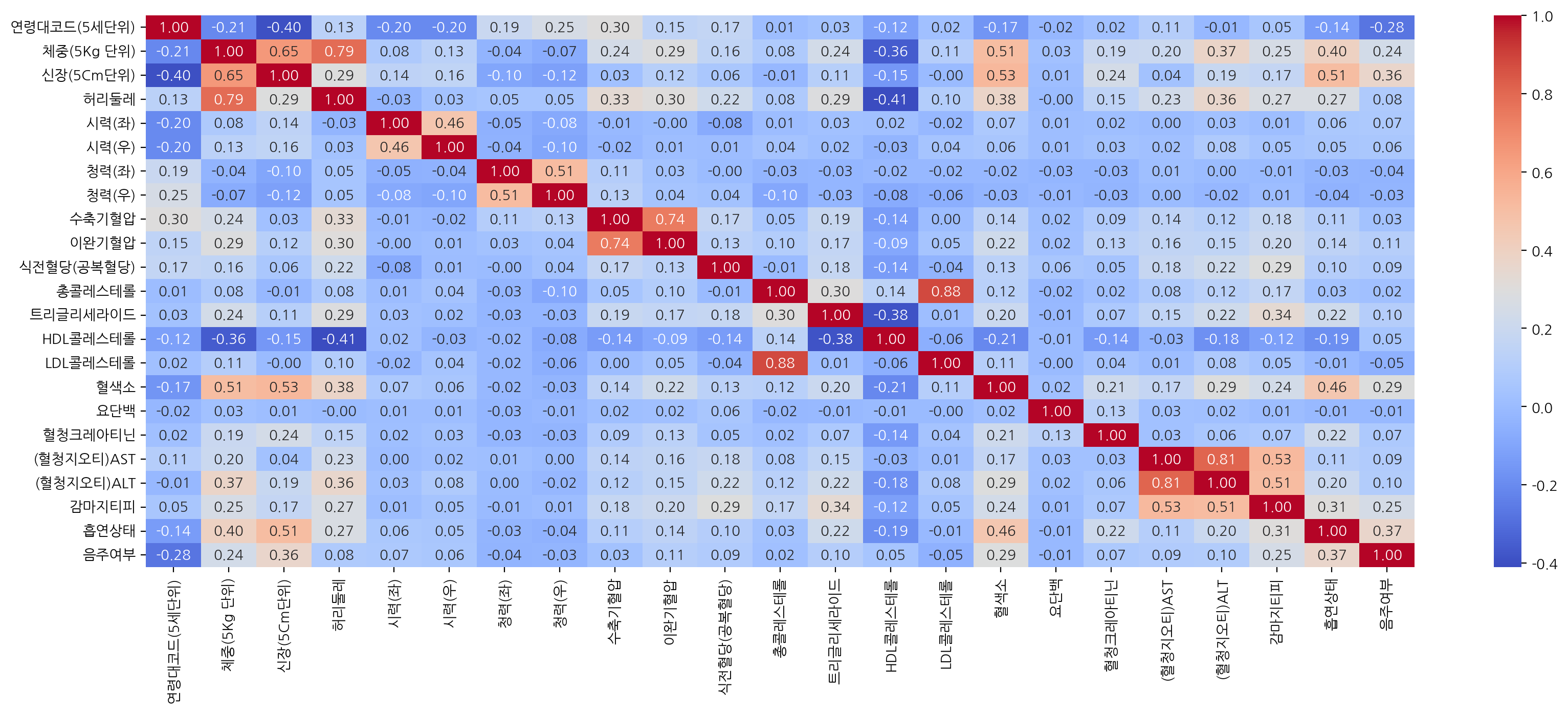
numpy의 np.triu 함수를 통해 상삼각형(위쪽 삼각형)을 마스킹하여 하삼각형(아래 삼각형)만을 출력할 수 있다.
mask = np.triu(np.ones_like(df_corr, dtype=np.bool))
plt.figure(figsize=(20, 7))
sns.heatmap(df_corr, annot=True, fmt=".2f", cmap="coolwarm", mask=mask)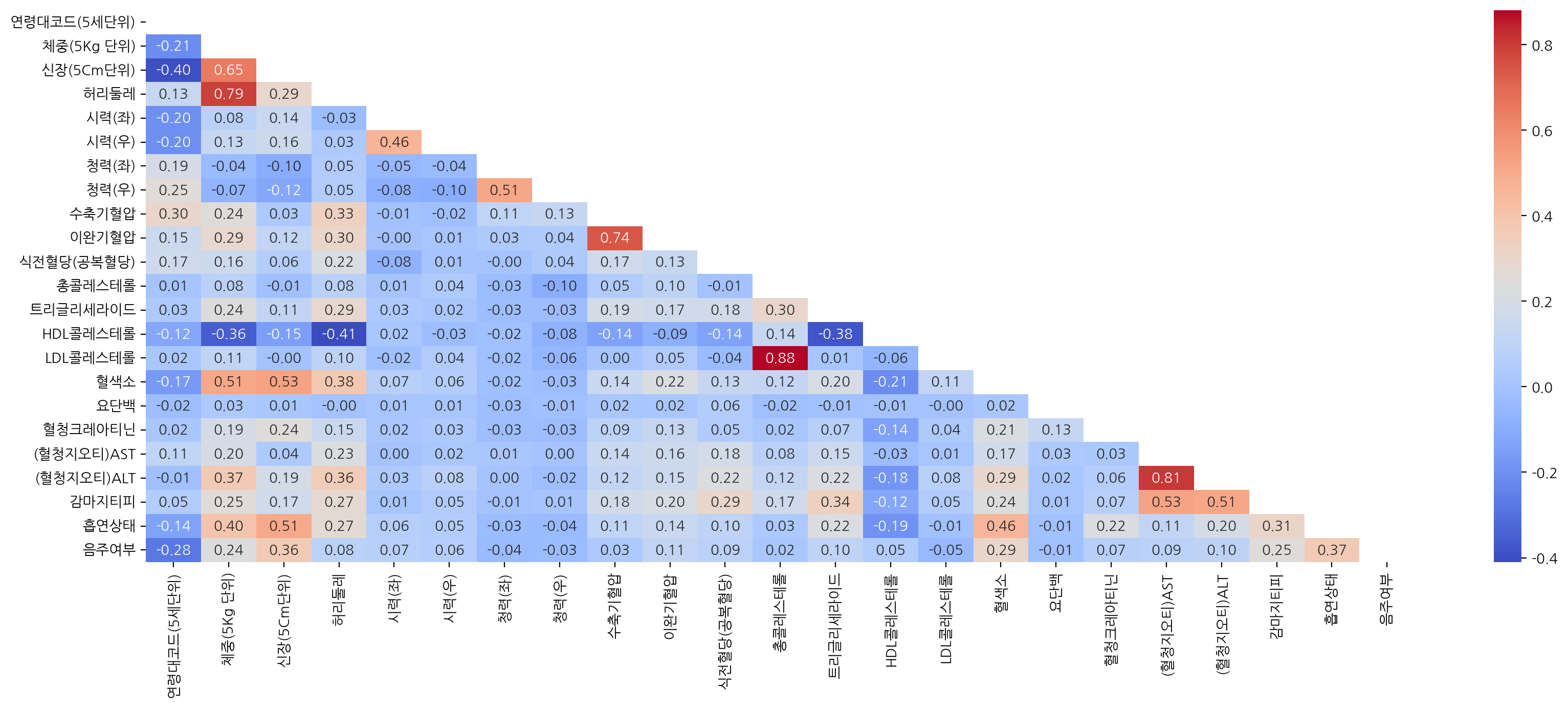
'Data Analysis & Viz' 카테고리의 다른 글
| [데이터분석] 대구 교통사고 데이터 분석하기 (0) | 2023.12.12 |
|---|---|
| [데이터분석] 온라인 화장품 해외 판매 분석하기 (0) | 2023.10.29 |
| [데이터시각화] Jupyter, Colab에서 matplotlib 한글 폰트 설정방법 (0) | 2023.10.15 |
| [데이터분석] 서울 종합병원 분포 확인하기 (0) | 2023.10.12 |
| [데이터분석] 엔트리를 통한 데이터 분석 (0) | 2023.09.01 |



